正交偏最小二乘判别分析(Orthogonal PLS-DA,OPLS-DA )是一种有监督的判别分析方法,是多变量统计分析方法,常用于代谢组学分析。本文详细记录了使用SMICA14.1软件进行OPLS-DA分析的图文步骤,希望能对初学者能有所帮助。视频学习可以参考b站论文讲解(香气物质、内含物质看这里)与SIMCA如何做OPLS-DA分析和Simca软件PCA OPLS-DA功能使用教程。
先放下载链接:SMICA14.1:https://pan.baidu.com/s/1fOIXZWEe3W36vau6pXnriw?pwd=9527 提取码:9527
注意OPLS-DA通常用于两组样品的对比分离,寻找差异物质,两组以上可考虑偏最小二乘法判别分析(PLS-DA)或者主成分分析(PCA)

导入数据
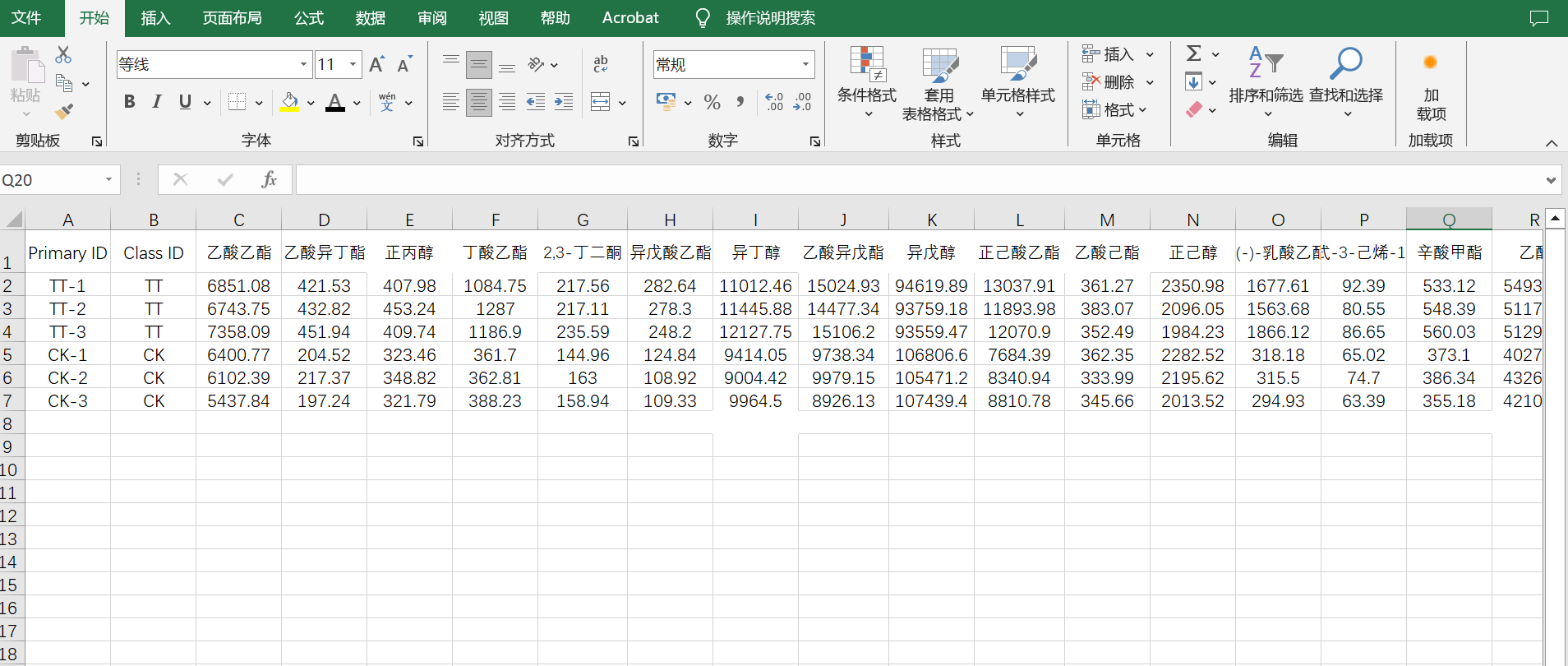
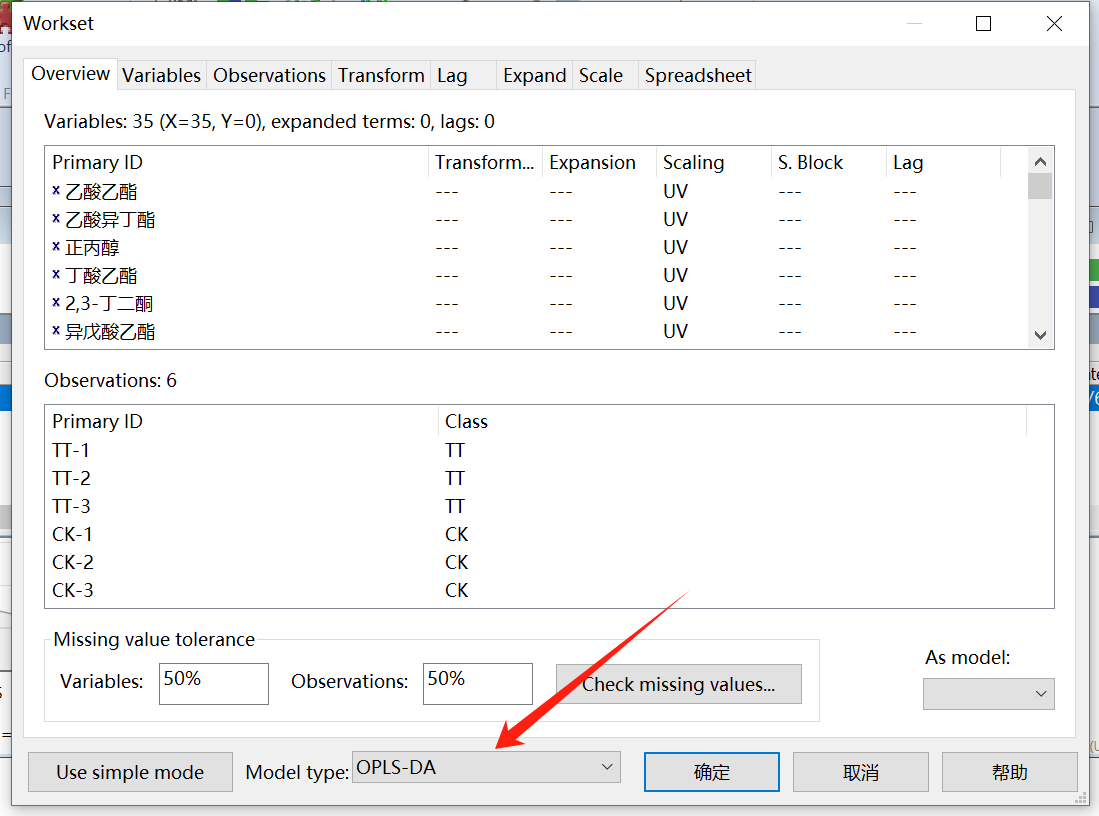
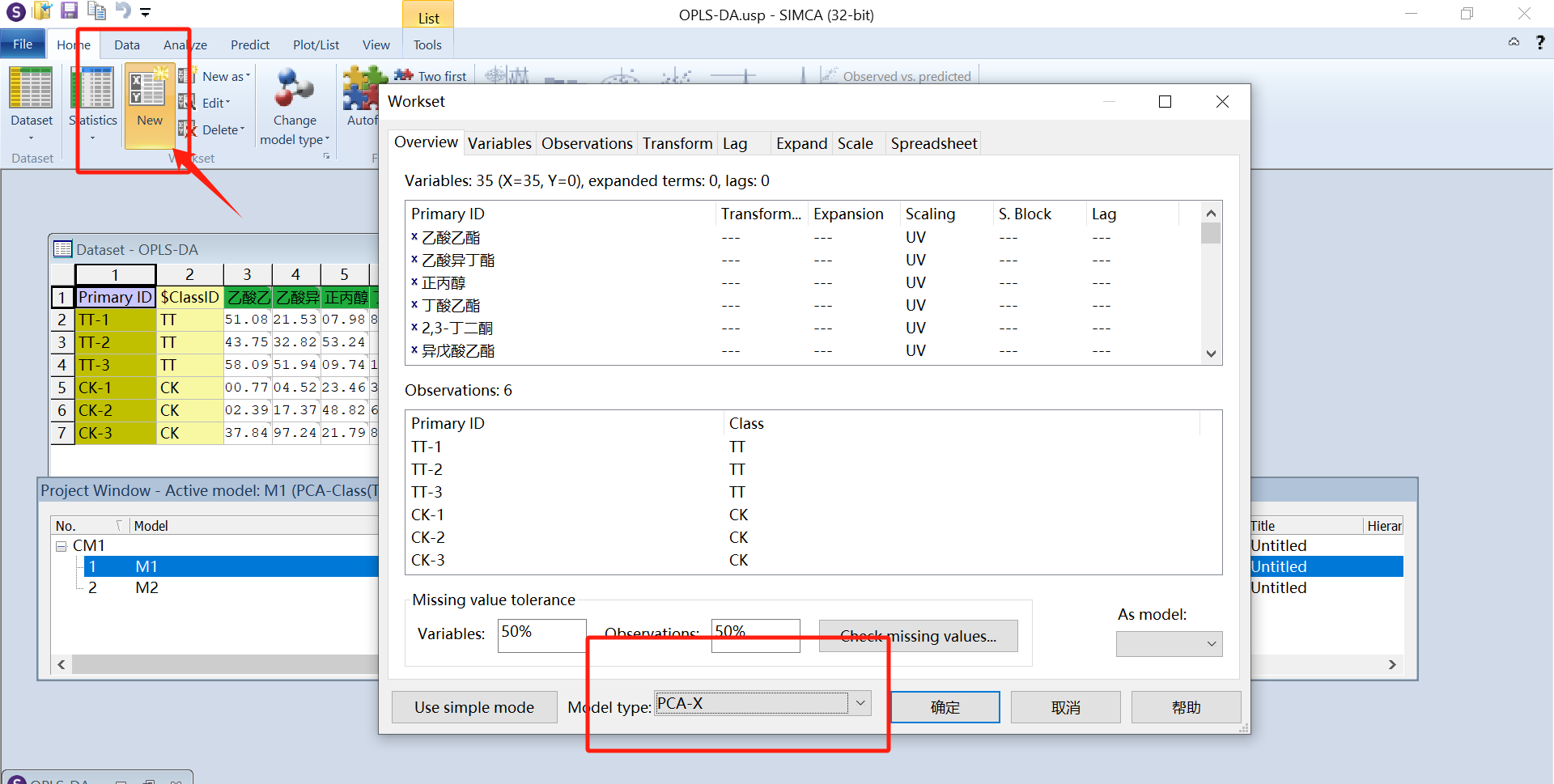
如图在excel表中列好数据,第一列Primary ID 为样品名称,第二列Class ID为样品组的分类,然后是各项指标(省的在软件里面设置分组了)。
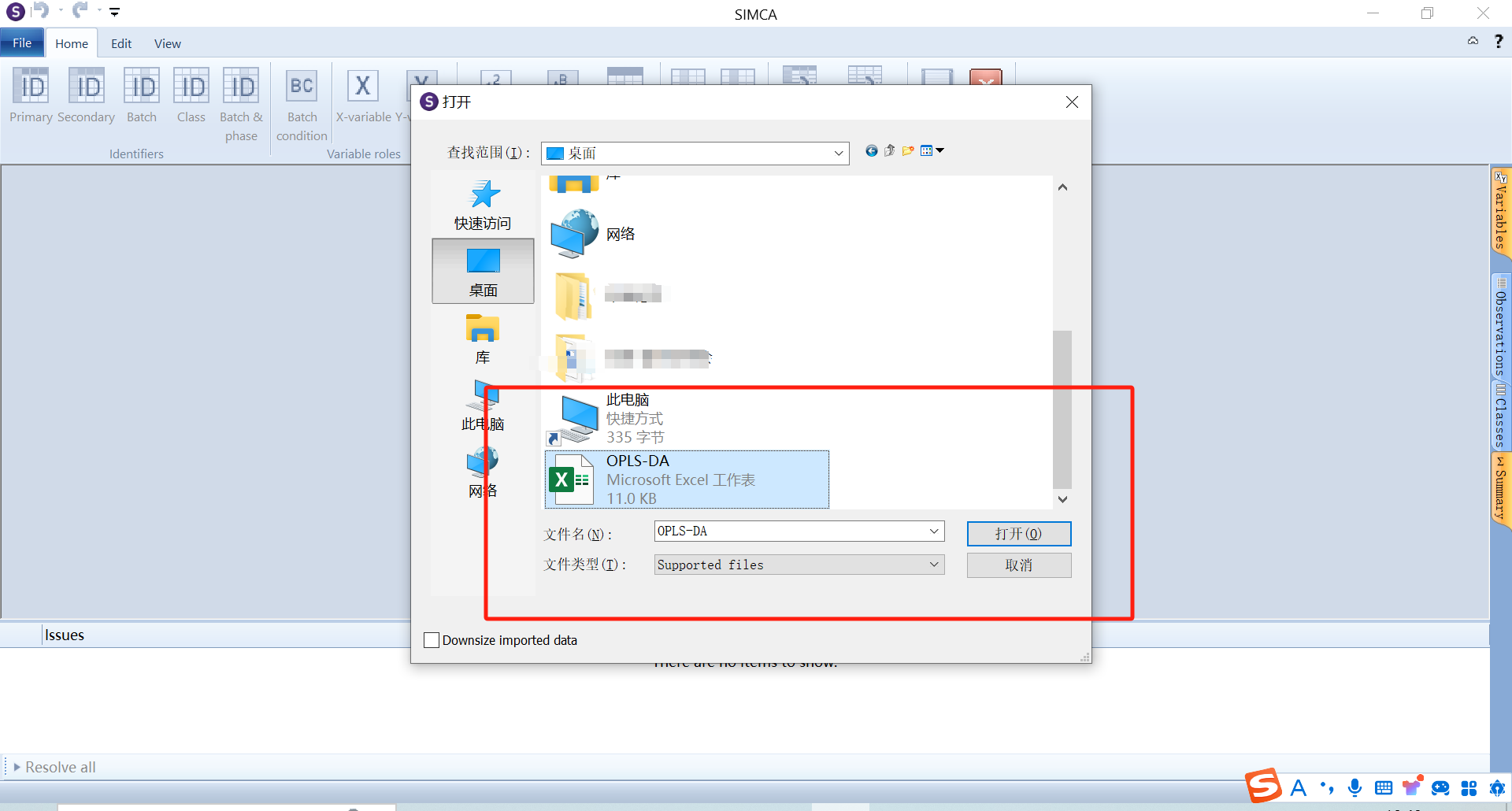
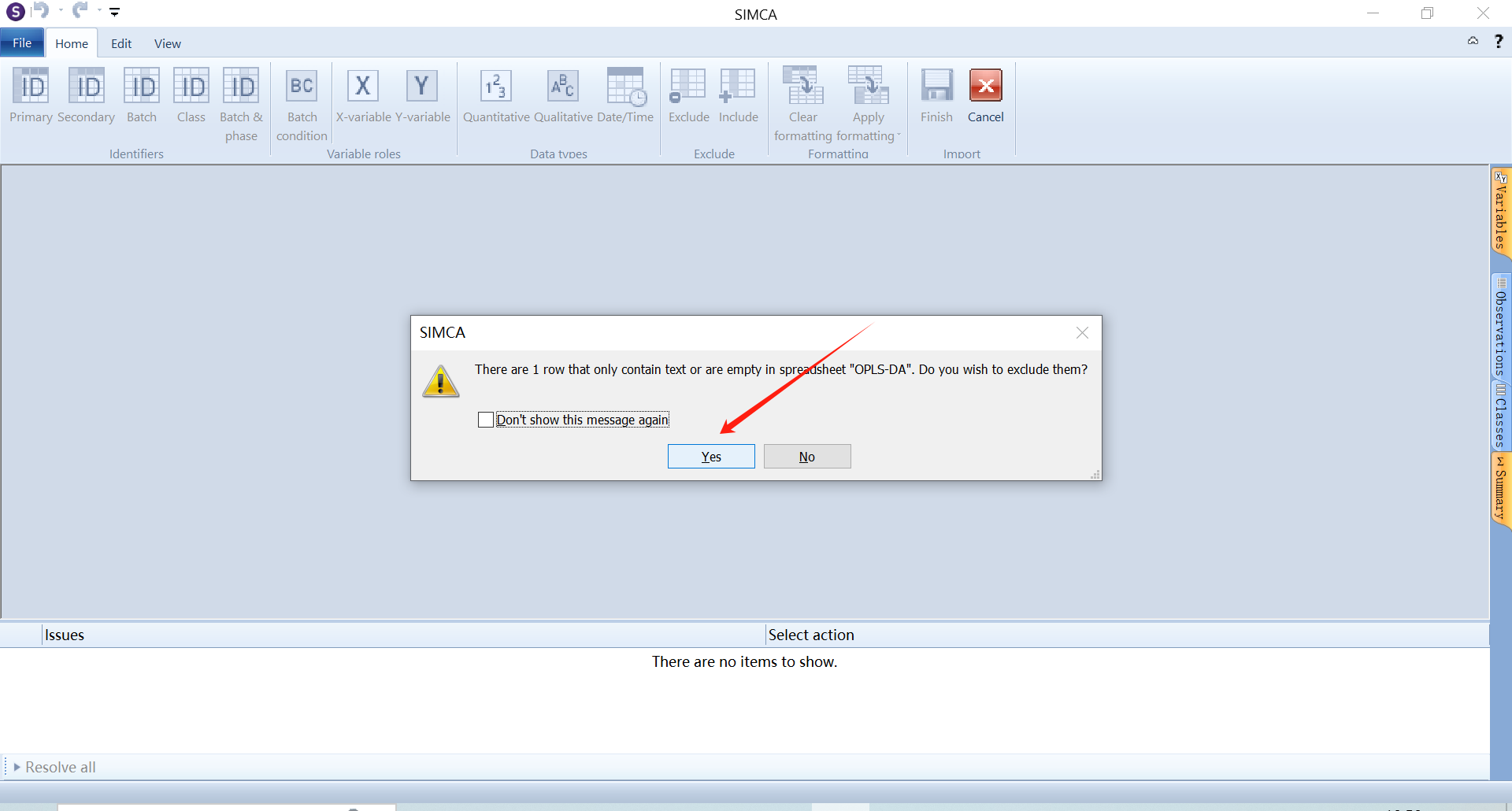
打开smica软件,将excel的数据导入,新建项目文件,然后保存。注意样本数据的缺失值(值为0)不能太多,当样本中的缺失值过多时,样本本身就缺乏了统计学意义,并且极有可能成为异常样本,可以考虑对缺失值过多的样本数据进行去除处理。当然也可以选择不排除这些缺失项,这些缺失项在最终的VIP值都均会大于1,表现为差异物质。
 |  |
 |  |
 |  |
数据分析
打开刚保存的项目文件,点击dataset即可看到原始数据。
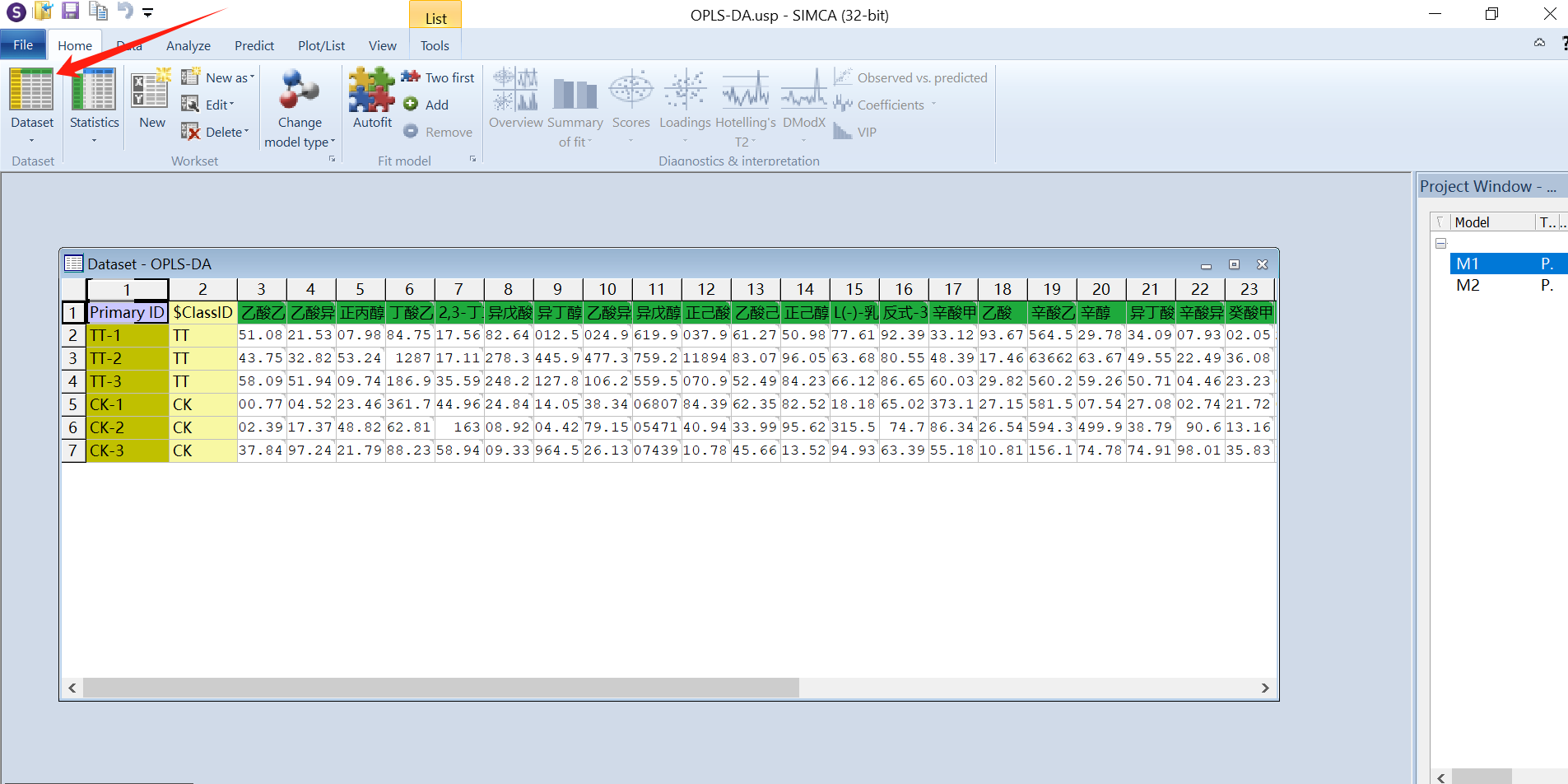
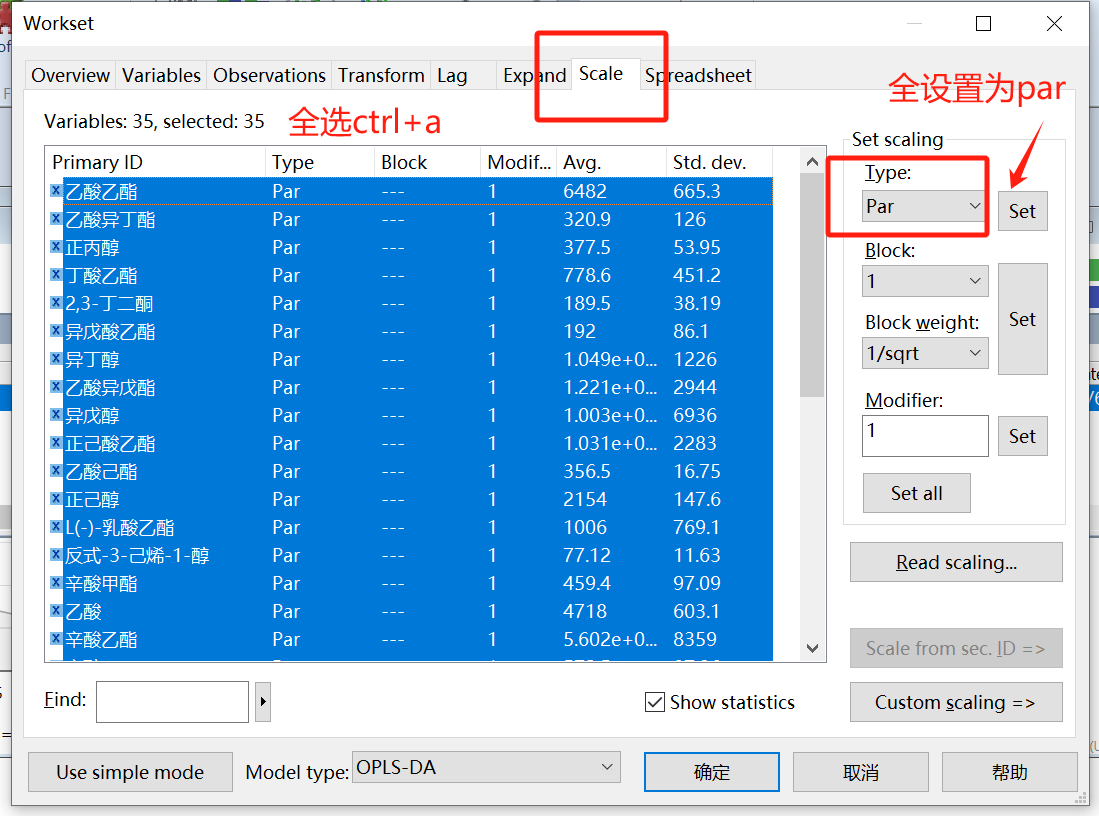
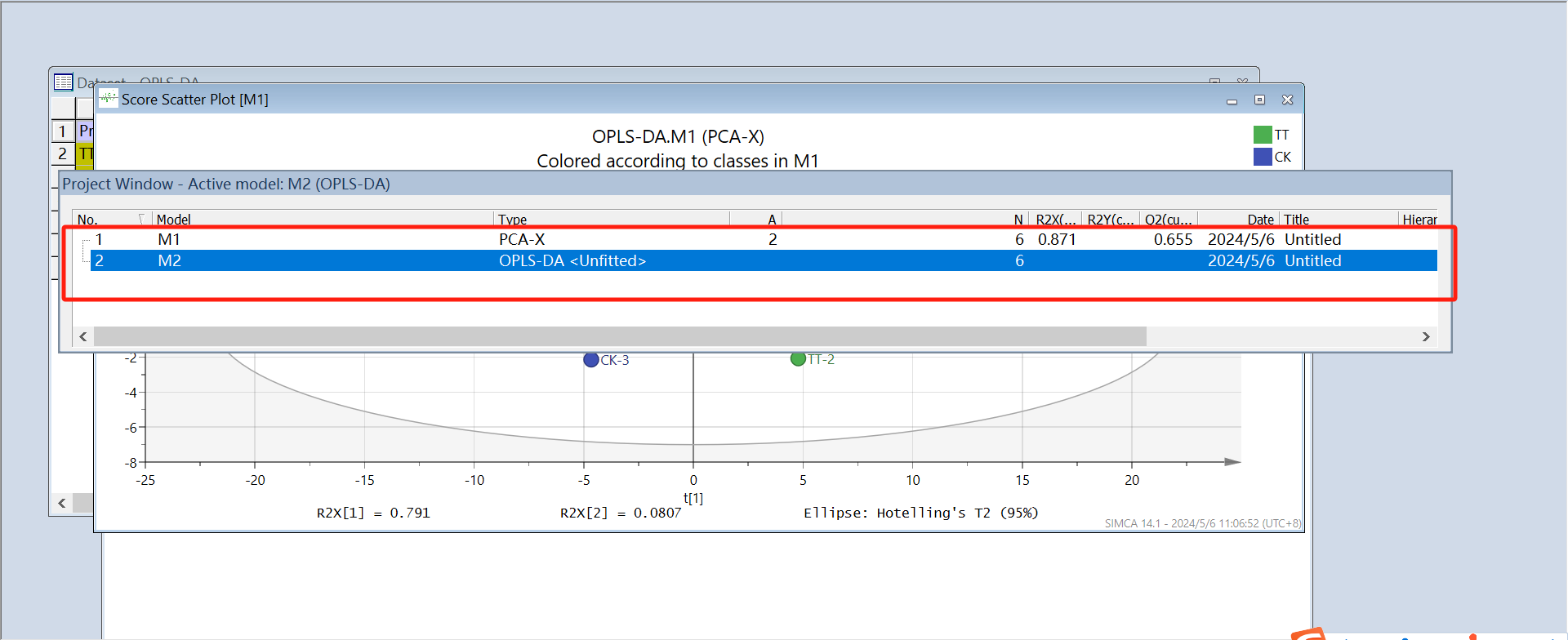
在进行OPLS-DA分析前,顺便学习下PCA(主成分分析)的操作。先选中CM1项目,点击上方菜单栏的“new”,这里我们选择Scale方式为Par,下方“model type”选择“PCA-X”,确定完成分析。Ctr是将原数据转化成离原点更近的新数据,Ctr=x-x拔;UV是将所有变量拥有同等的重要性,UV=x-x拔/SD;Par相比UV更接近原始测量数据,Par=x-x拔/根号SD,可以根据数据情况选择合适的Scale方式。
|
| |
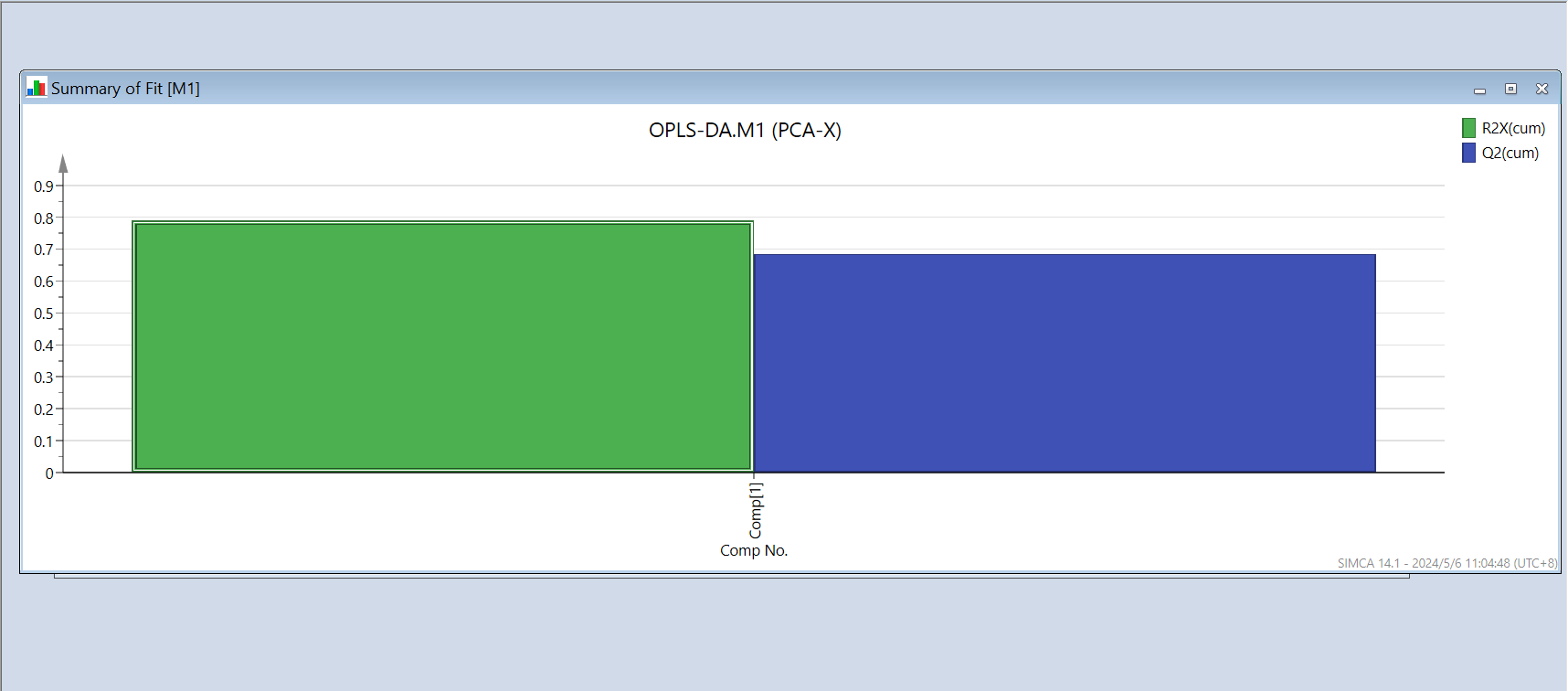
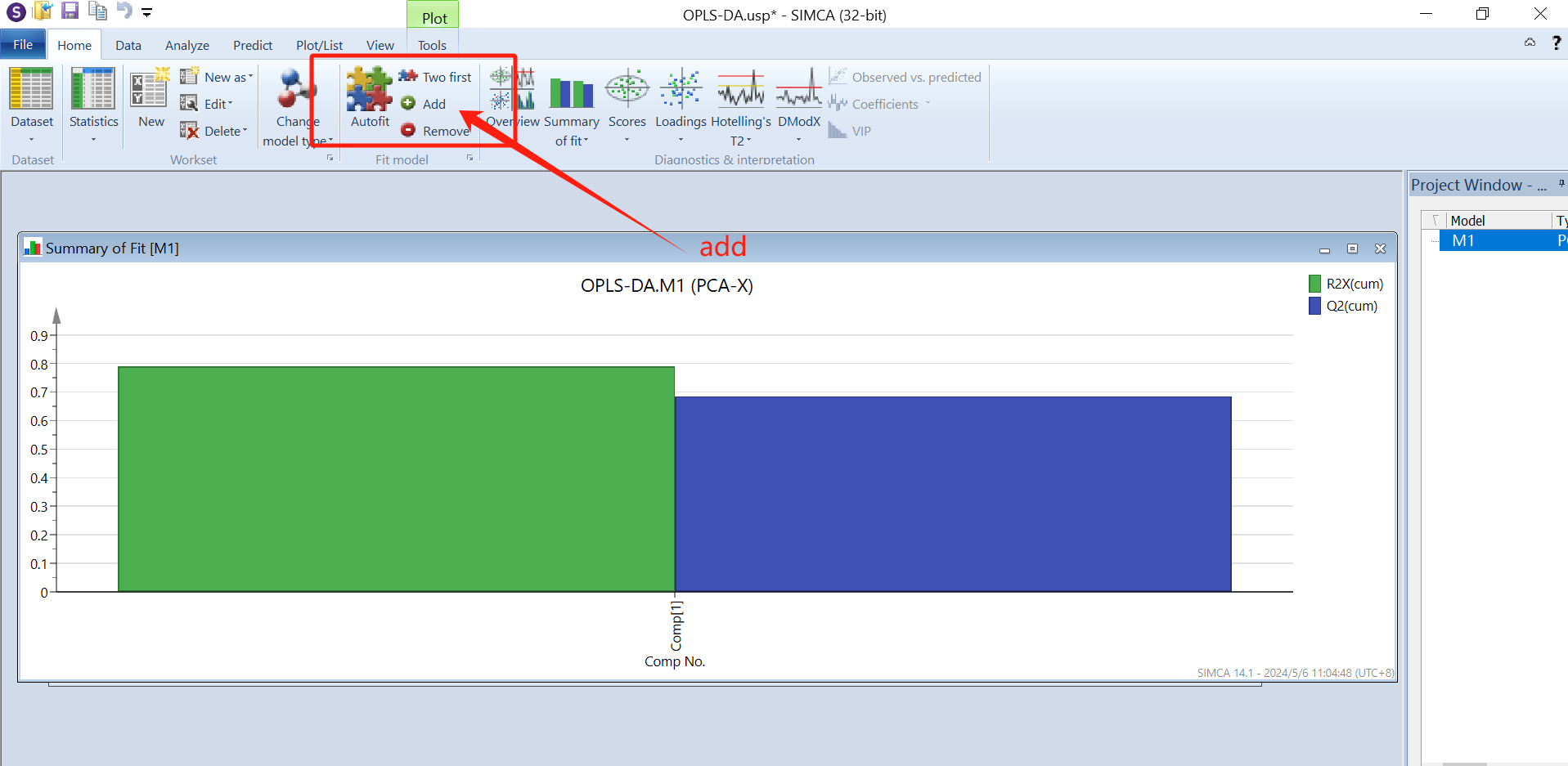
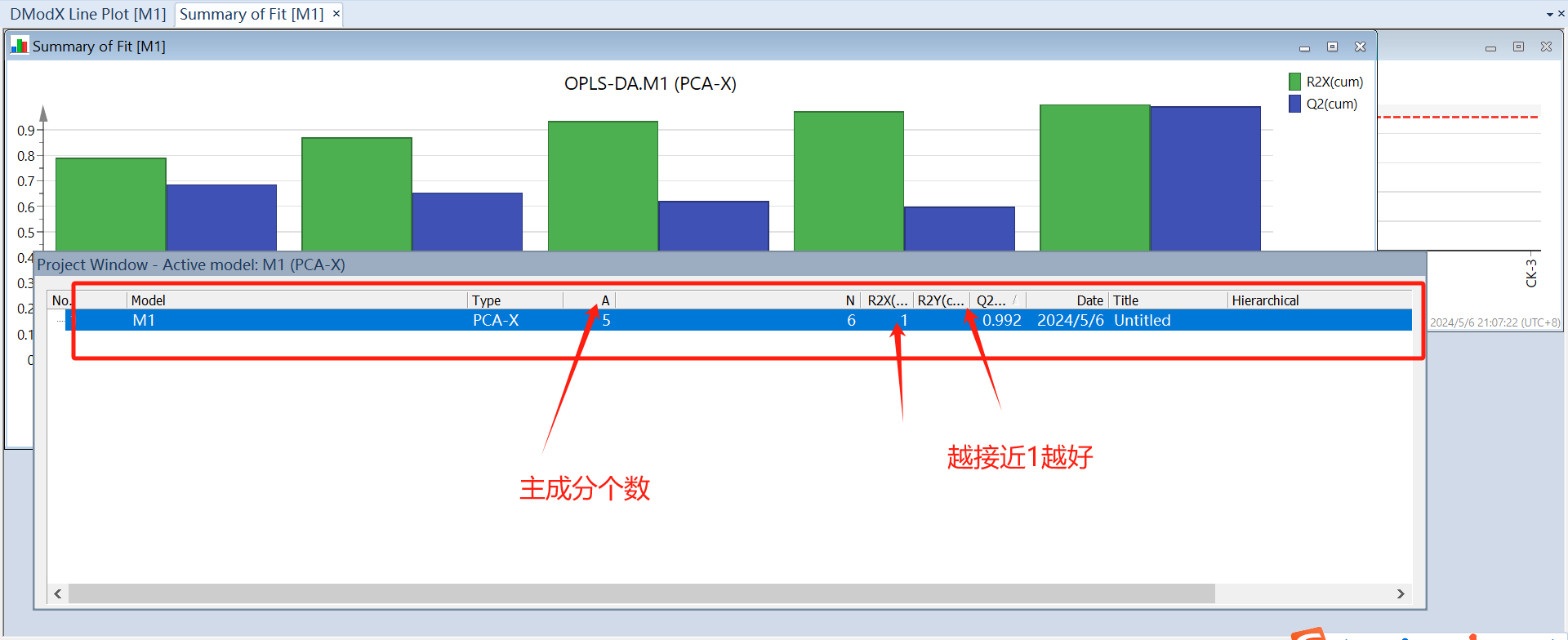
分析完会弹出一个对话框M1(PCA-X),即交叉验证的结果,选中M1点击上方菜单栏的Autofit(自动拟合),则出现一个柱状图,点击“add”即增加一个主成分,这里根据R2X、R2Y和Q2的值来选择(add or remove)主成分的个数,最少为2个,且都要满足>0.5。
| 指标 | 全称与含义 | 衡量内容 | 数值解读 | 重要性 |
|---|---|---|---|---|
| R2X | X的解释率 | 模型对原始数据的概括能力 | 越高,拟合数据越好 | 中等,需结合其他指标看 |
| R2Y | Y的解释率 | 模型对分组差异的描述能力 | 越高,区分组别越好 | 高,模型的核心目标 |
| Q2 | 预测能力 | 模型对新样本的预测能力 | >0.5 较好,<0 模型无效 | 最高,评估模型可靠性的金标准 |
(表格来源于deepseek)
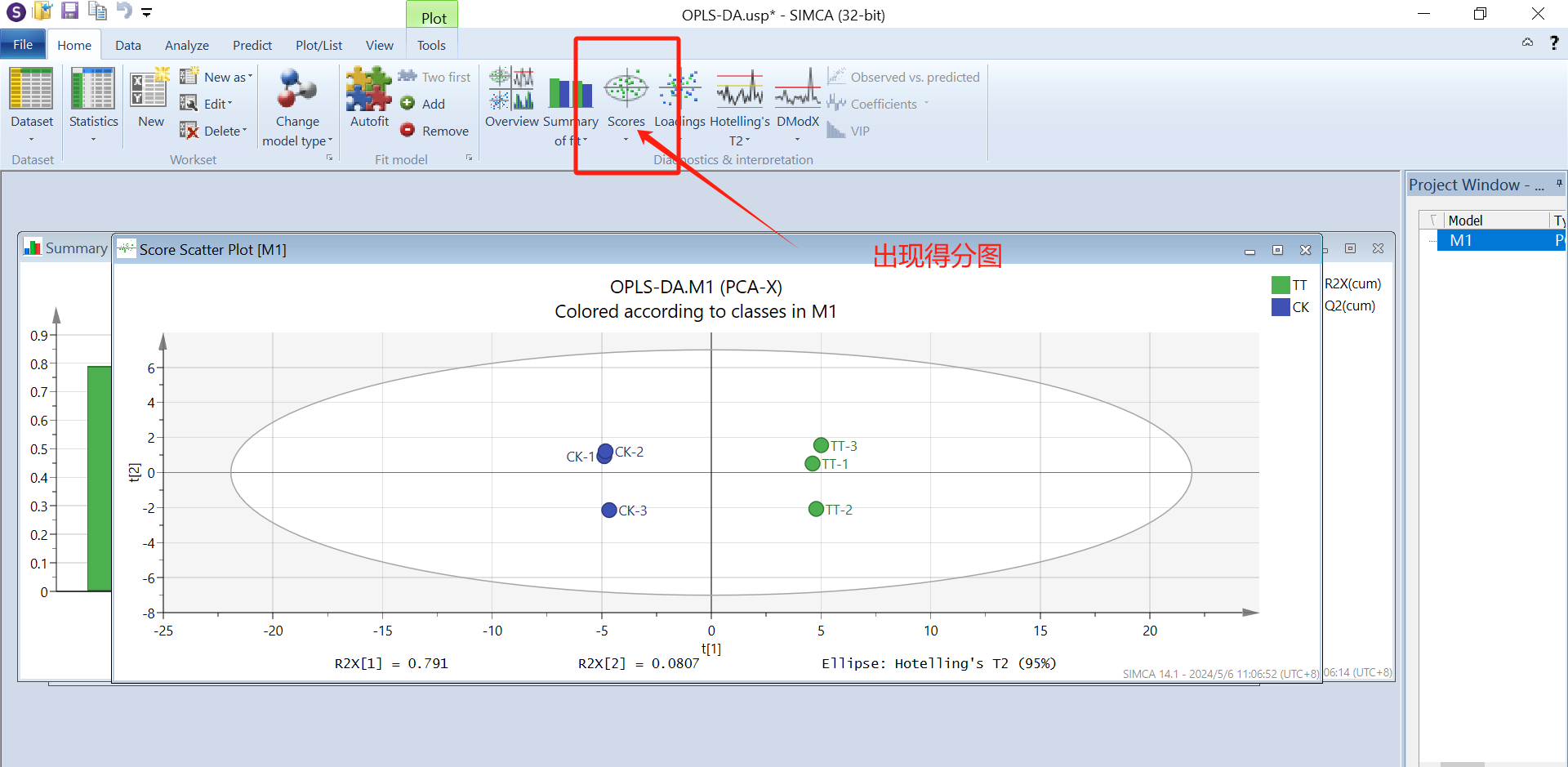
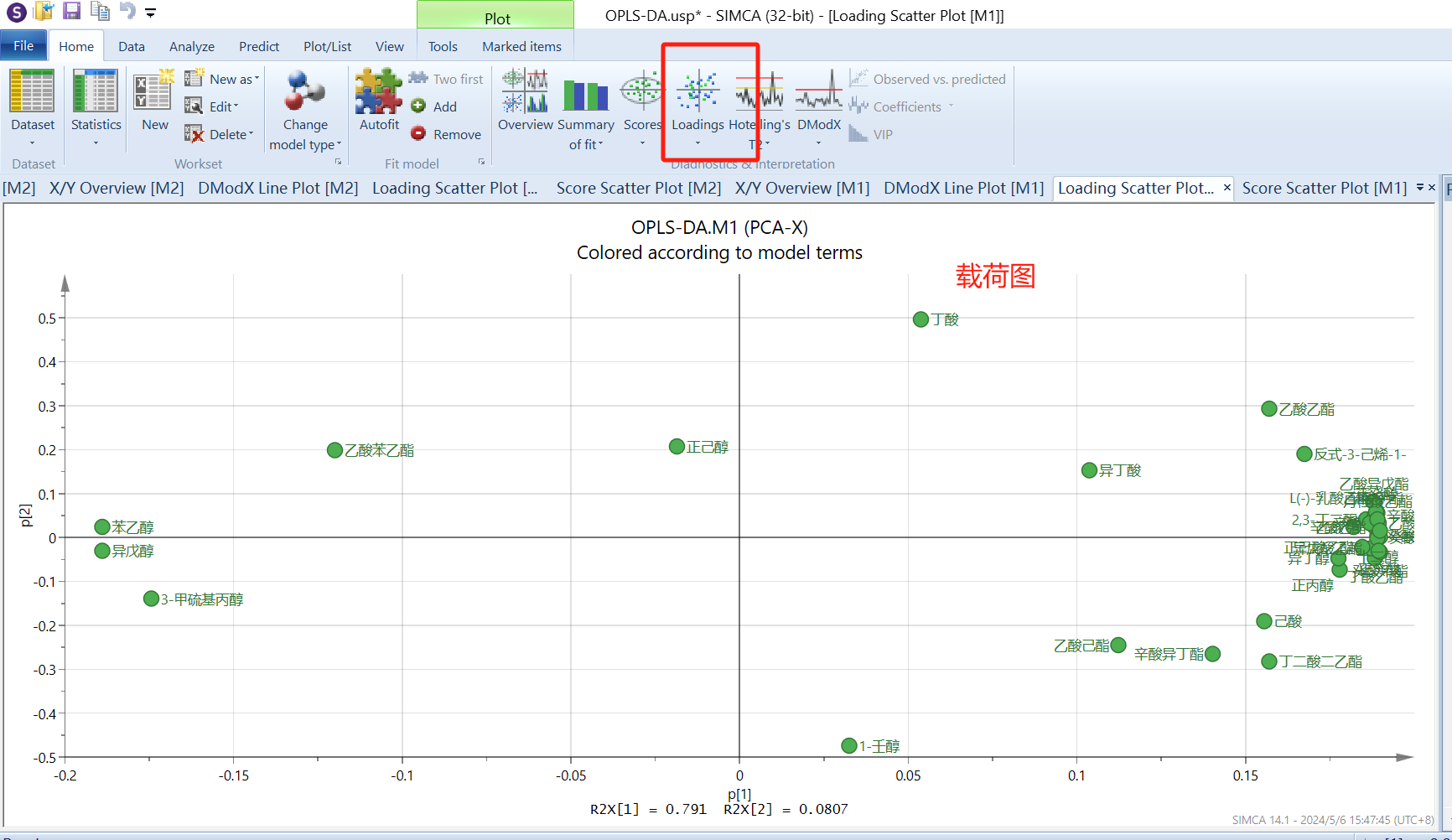
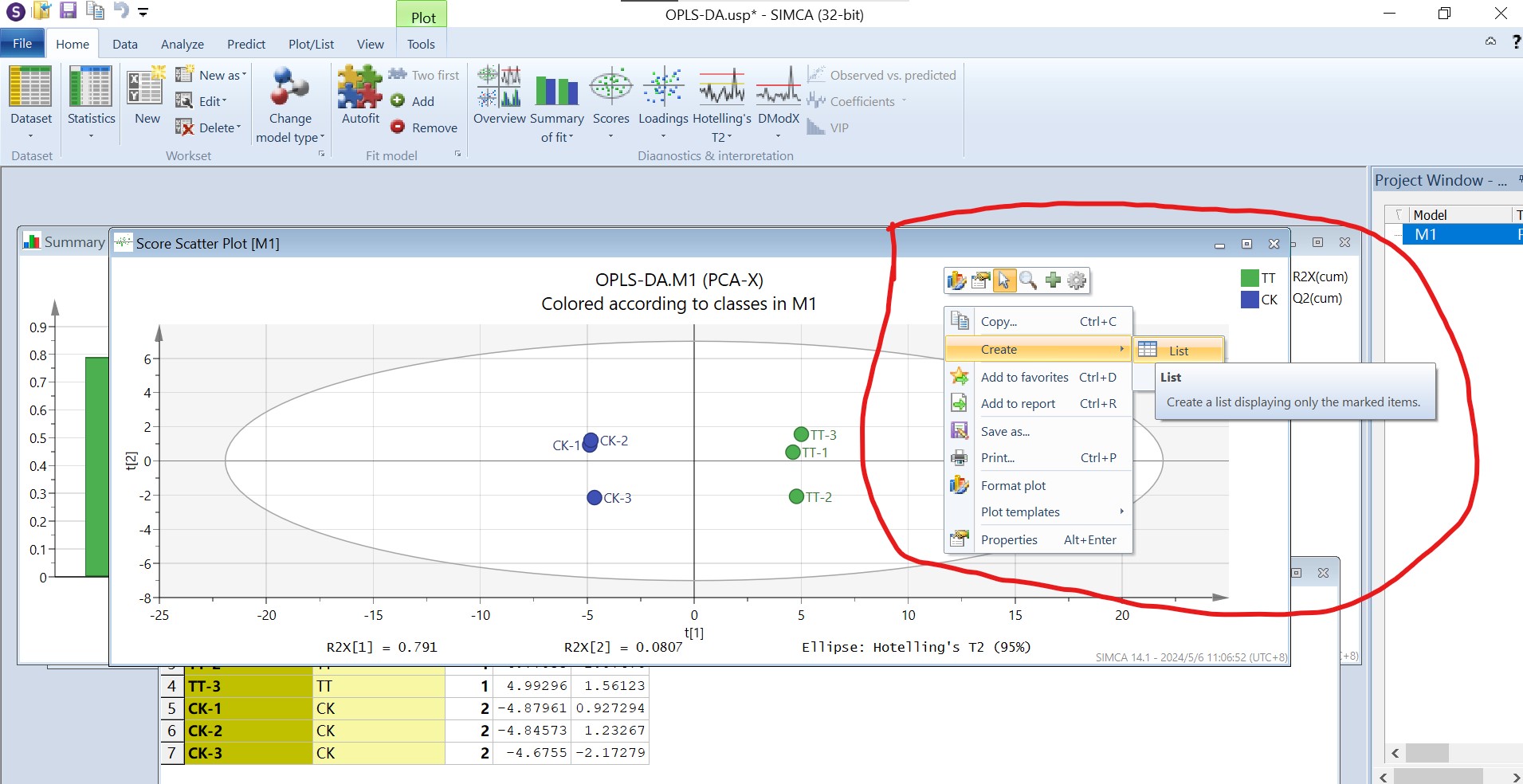
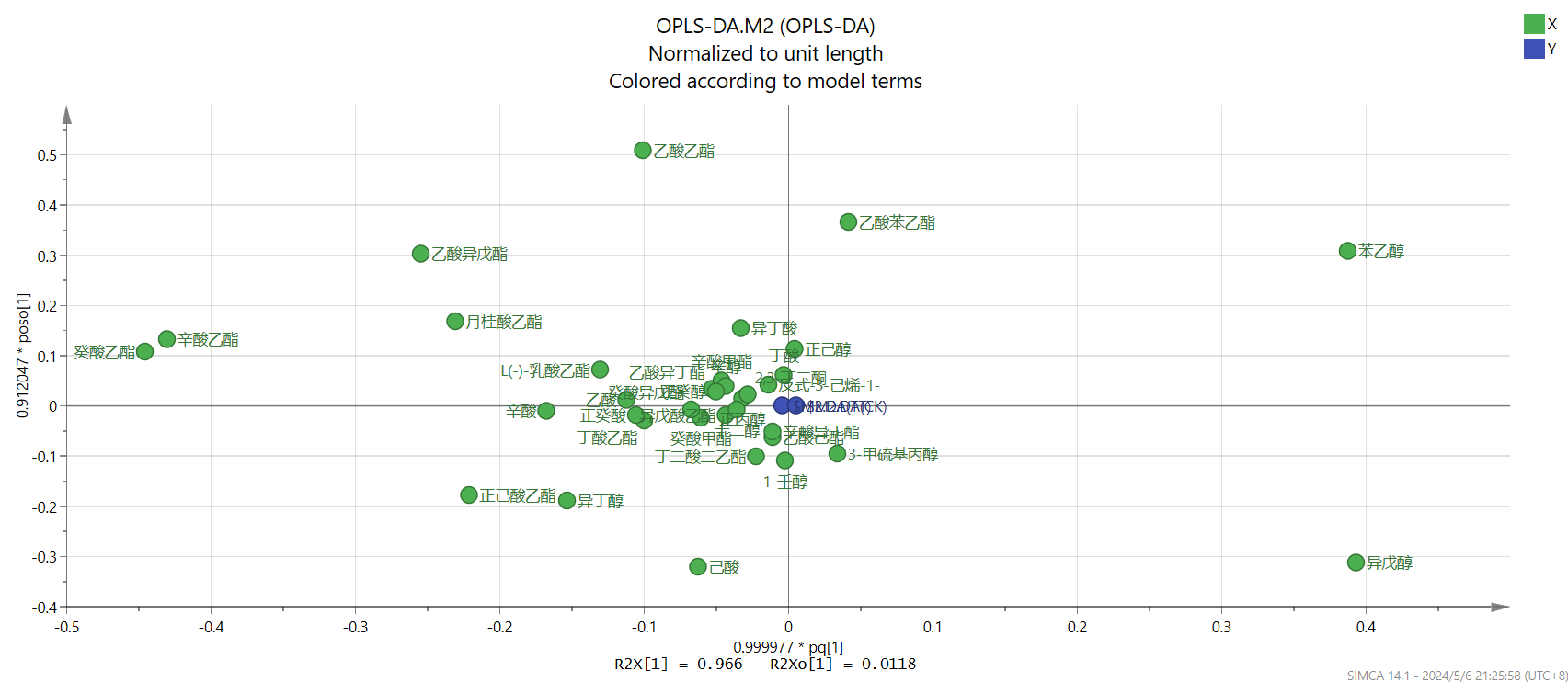
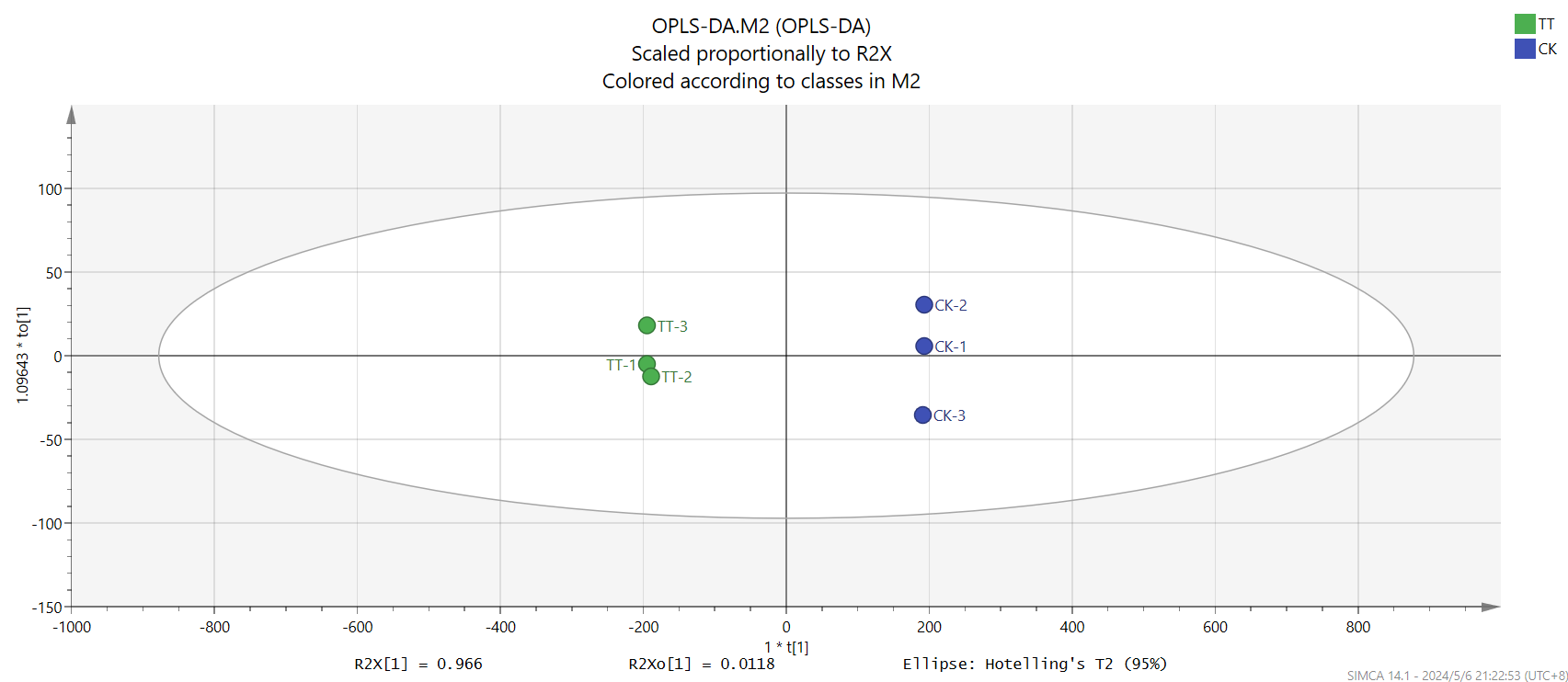
这里只选择了两个主成分,但也满足要求,然后点击“score”,产生一个得分图,点击loadings产生载荷图。右键点击“create”→”list”,可以导出得分图和载荷图的原始数据,可以在smica软件上进行图片的美化,也可以将这些数据导入到origin软件中进行作图,修改美化。
 |  |
 |  |
 |  |
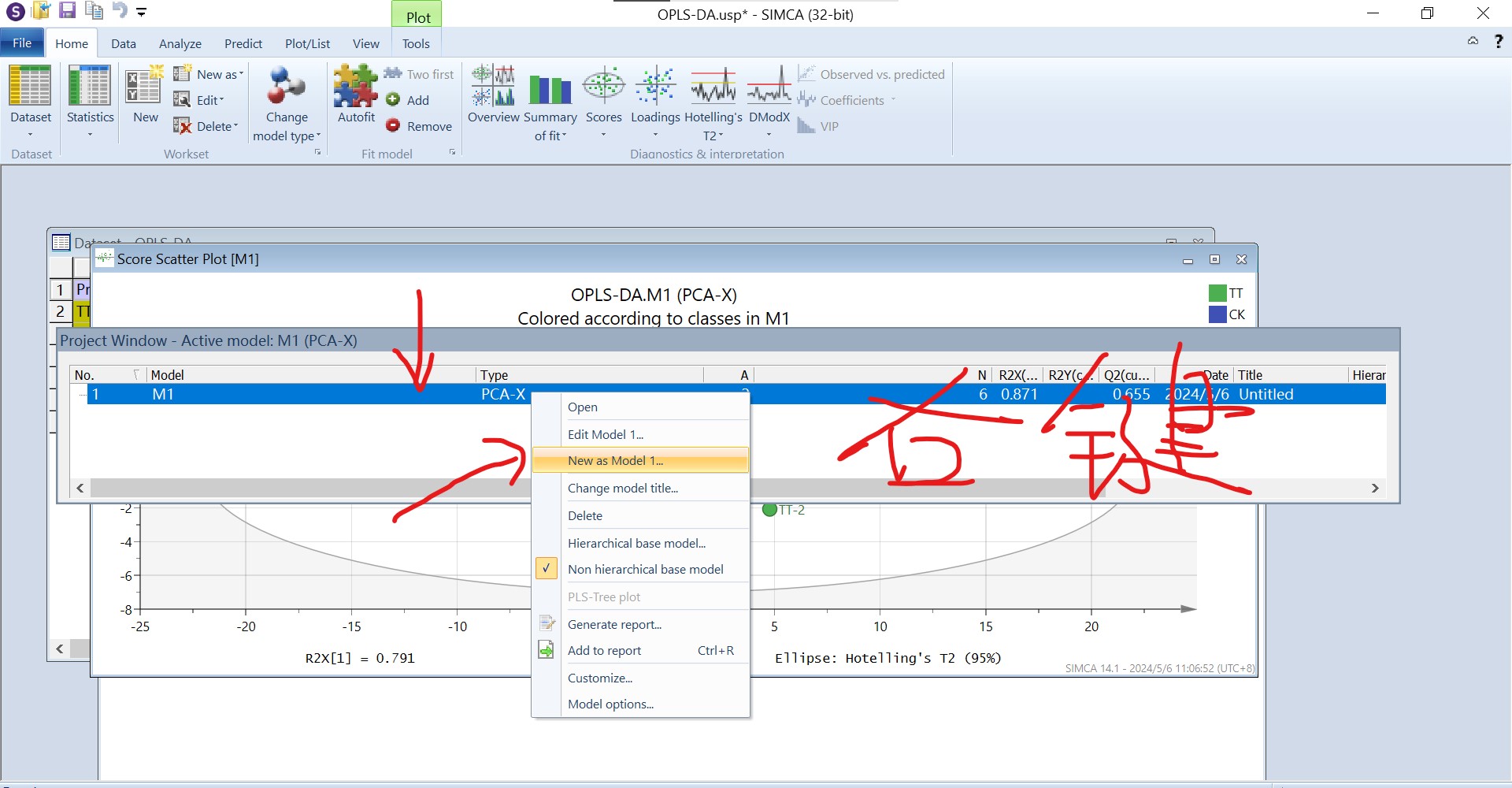
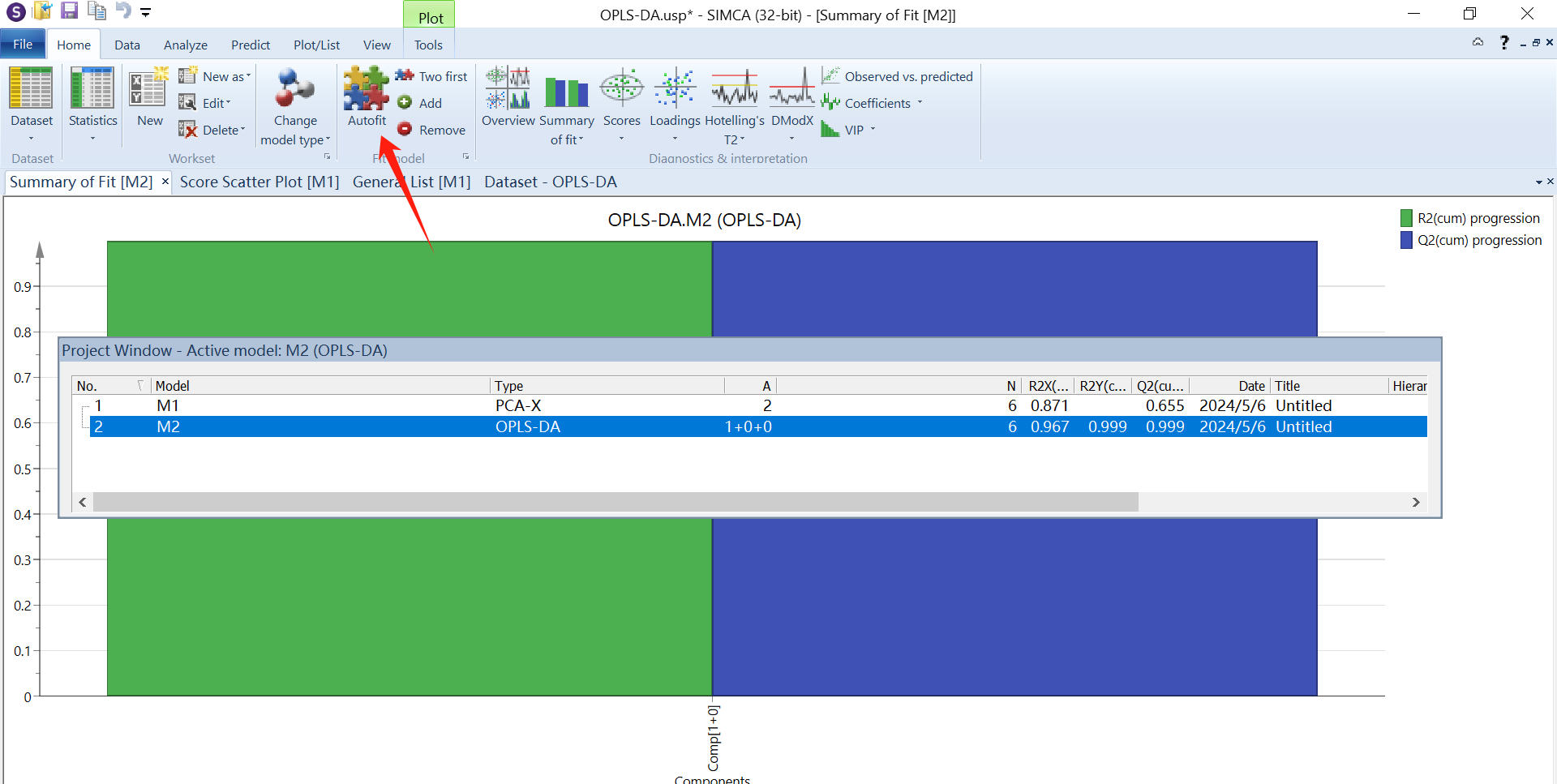
点击M1,右键选择“new as model 1”我们开始进行OPLS-DA、PLS-DA分析等操作
(想跳过PCA的,直接选中CM1,点击“new as model 1”,然后按照下面图片操作就行)
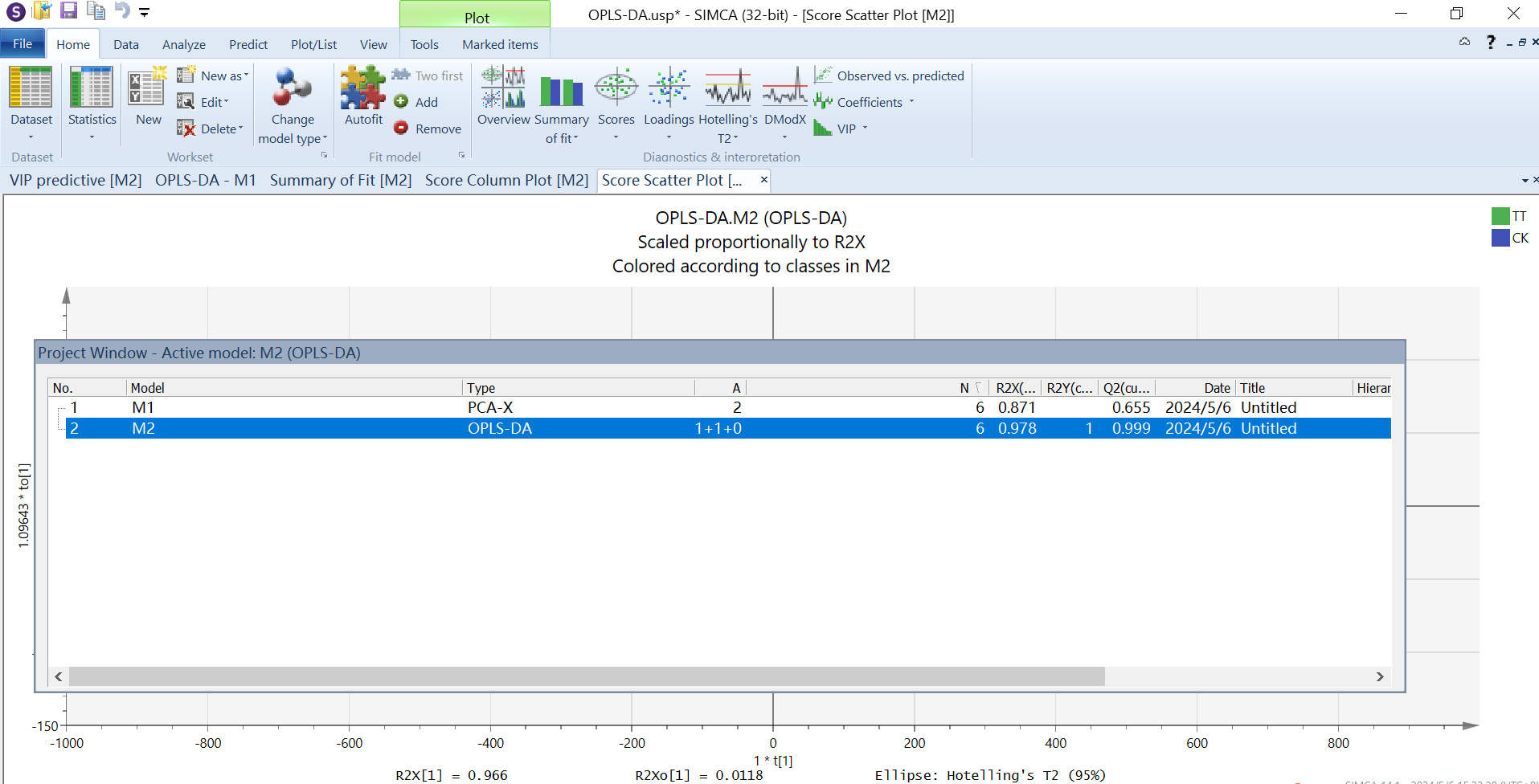
根据图片进行操作,“scale”中全设置为par(较UV而言,Par处理后模型对数据的敏感度更高),“model type”选择“OPLS-DA”,然后自动拟合,“add”一个主成分(这里仅两个主成分的R2Y和Q2值就很高),点击“score”和“loadings”生产得分图和载荷图。
得分图可以看两组样品是不是分得很开,存不存在差异,横坐标表示预测成分得分值,横坐标方向可以看出组间的差距;纵坐标表示正交成分得分值,纵坐标方向可以看出组内的差距;百分比表示成分对数据集的解释度。
载荷图可以看出成分的聚类程度,挨在一块的成分表示相似度高,能被聚成为一类;主成分的载荷代表变量与主成分之间的相关性(正相关和负相关),因此第一象限的点表现为强正相关,第四象限的点表现为强负相关。
 | |
 |  |
 |  |
 | 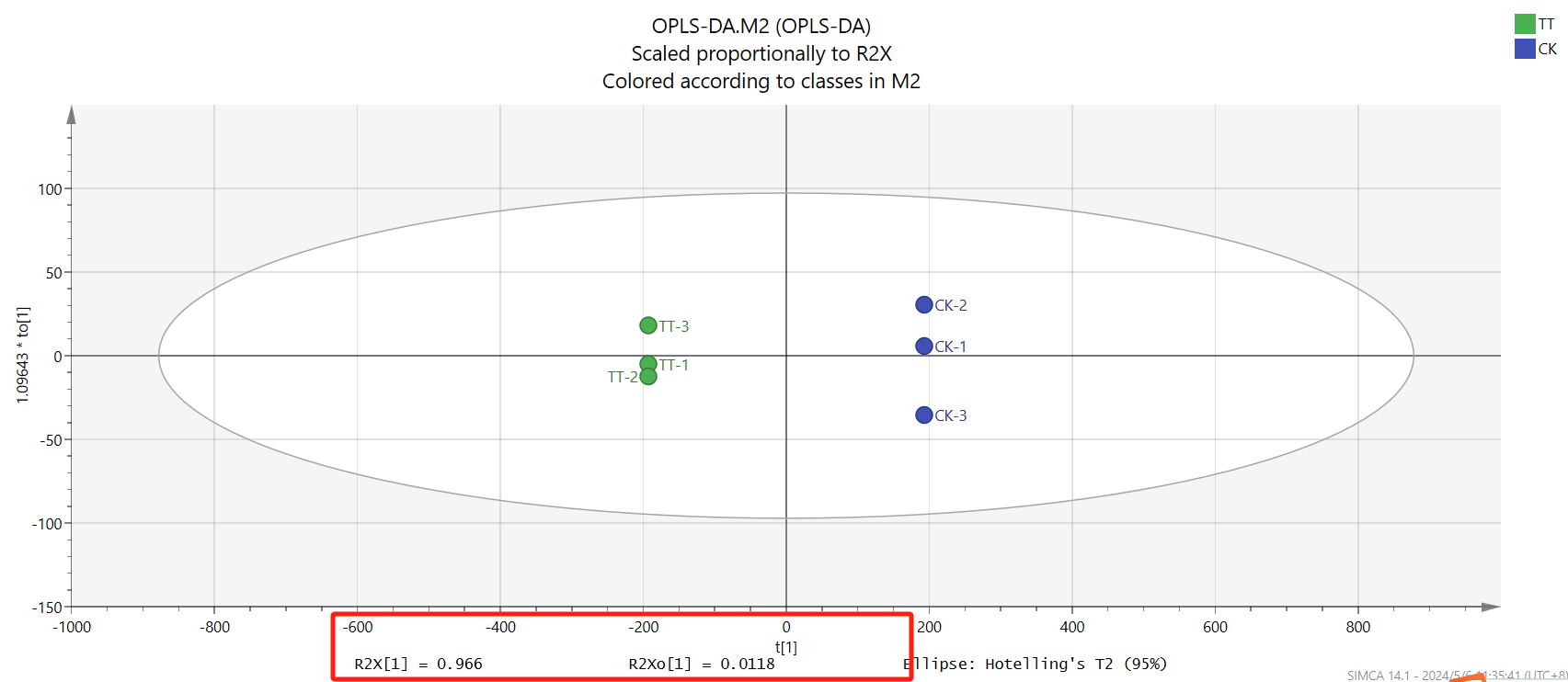 |
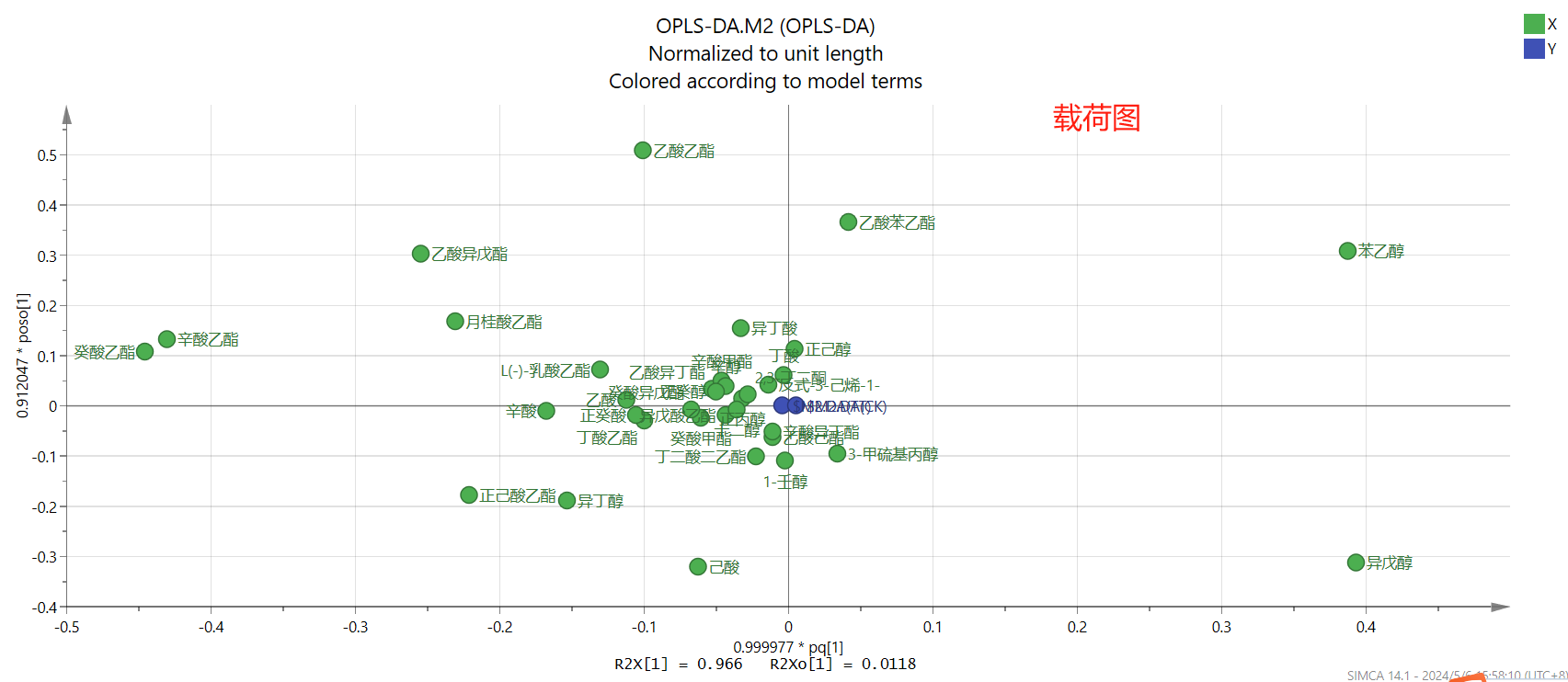 | |
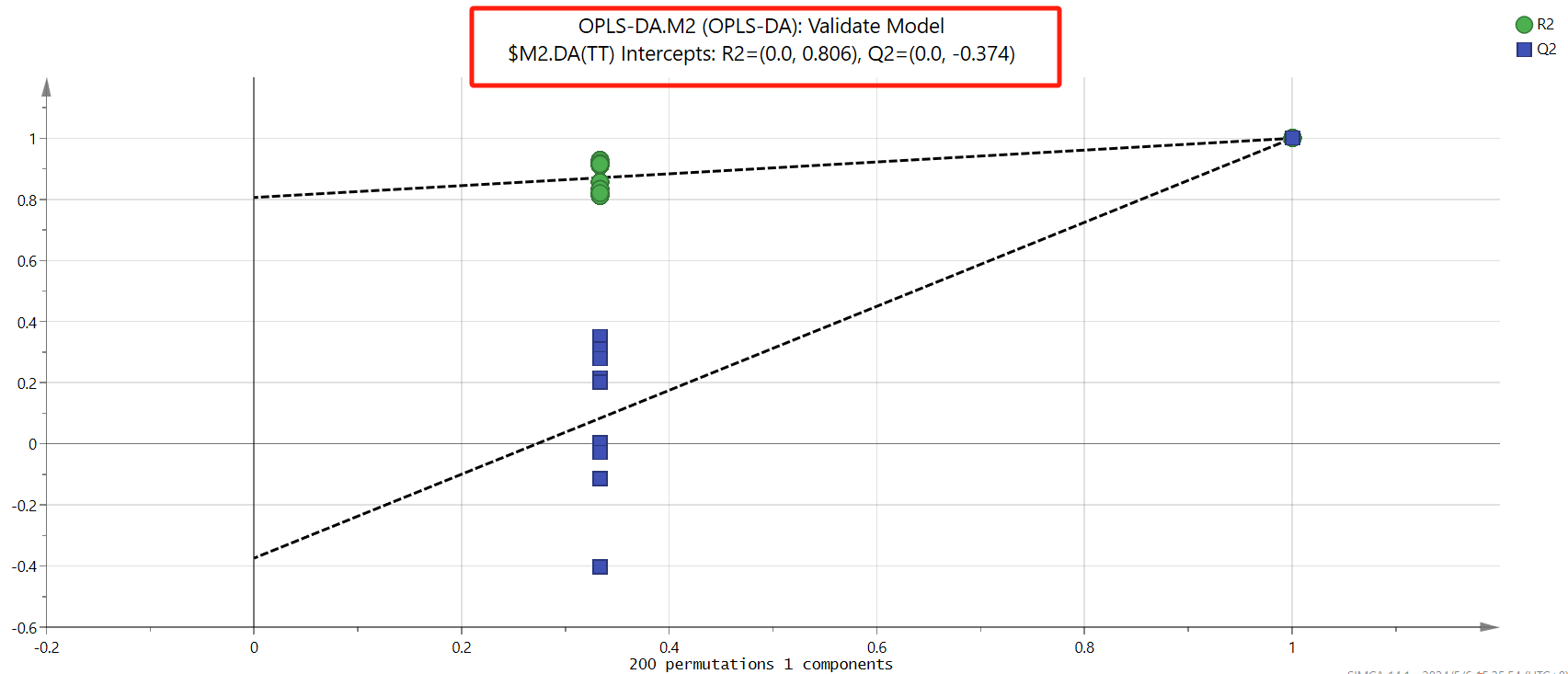
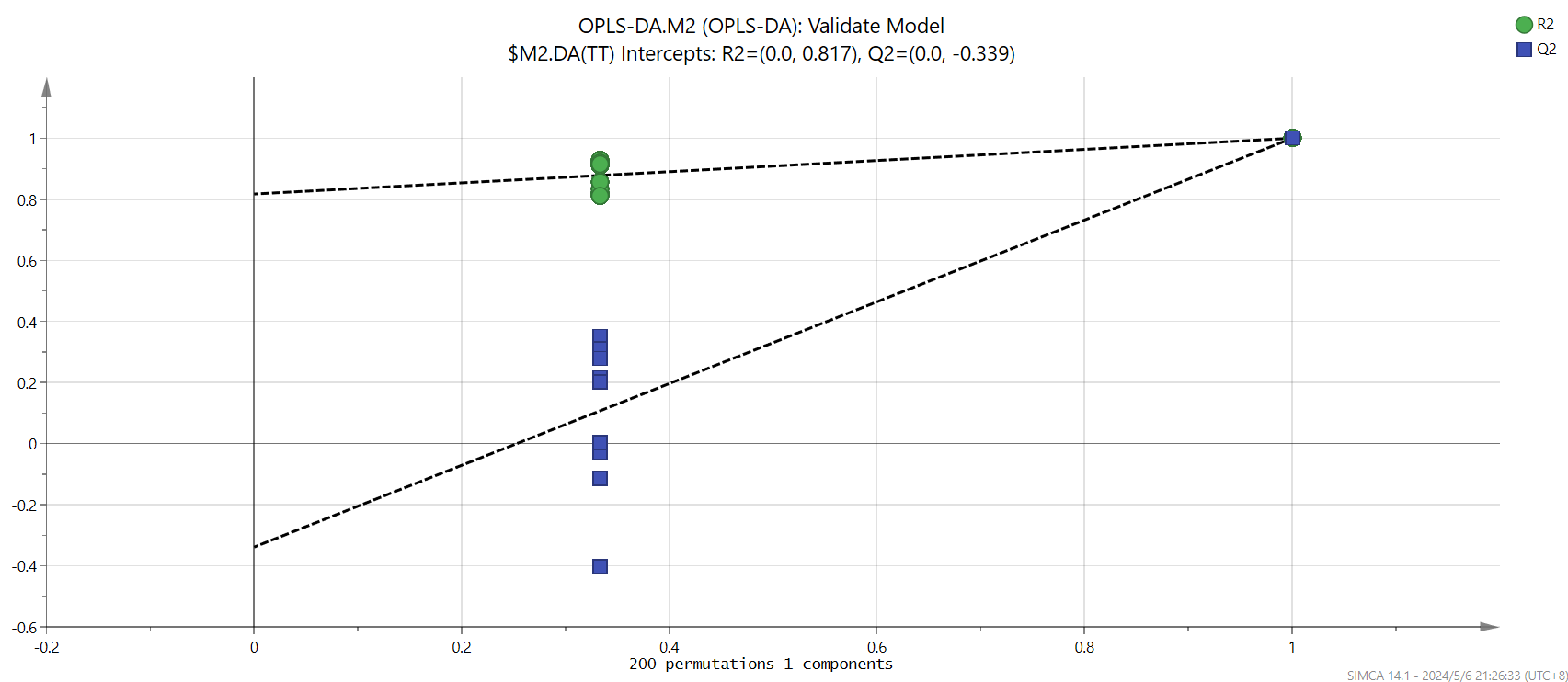
接着需要对OPLS-DA结果进行置换检验(permutation test),判断模型是否存在“过拟合”。选中项目M2,点击“analyze”界面的“permutations”,将20更改为“200”,然后确定。
判断条件:1. 原始的R2Y和Q2Y(最右边的两个点)总是大于左边那些置换后对应的值(左边那些散点)。2. 看截距,根据经验判断,优秀的模型R2Y的截距不超过0.3-0.4,Q2Y的截距不超过0.05(通常为负值)。但在实际项目中,能满足两条斜线的斜率为正,且Q2Y的截距不超过0.05就可以了。
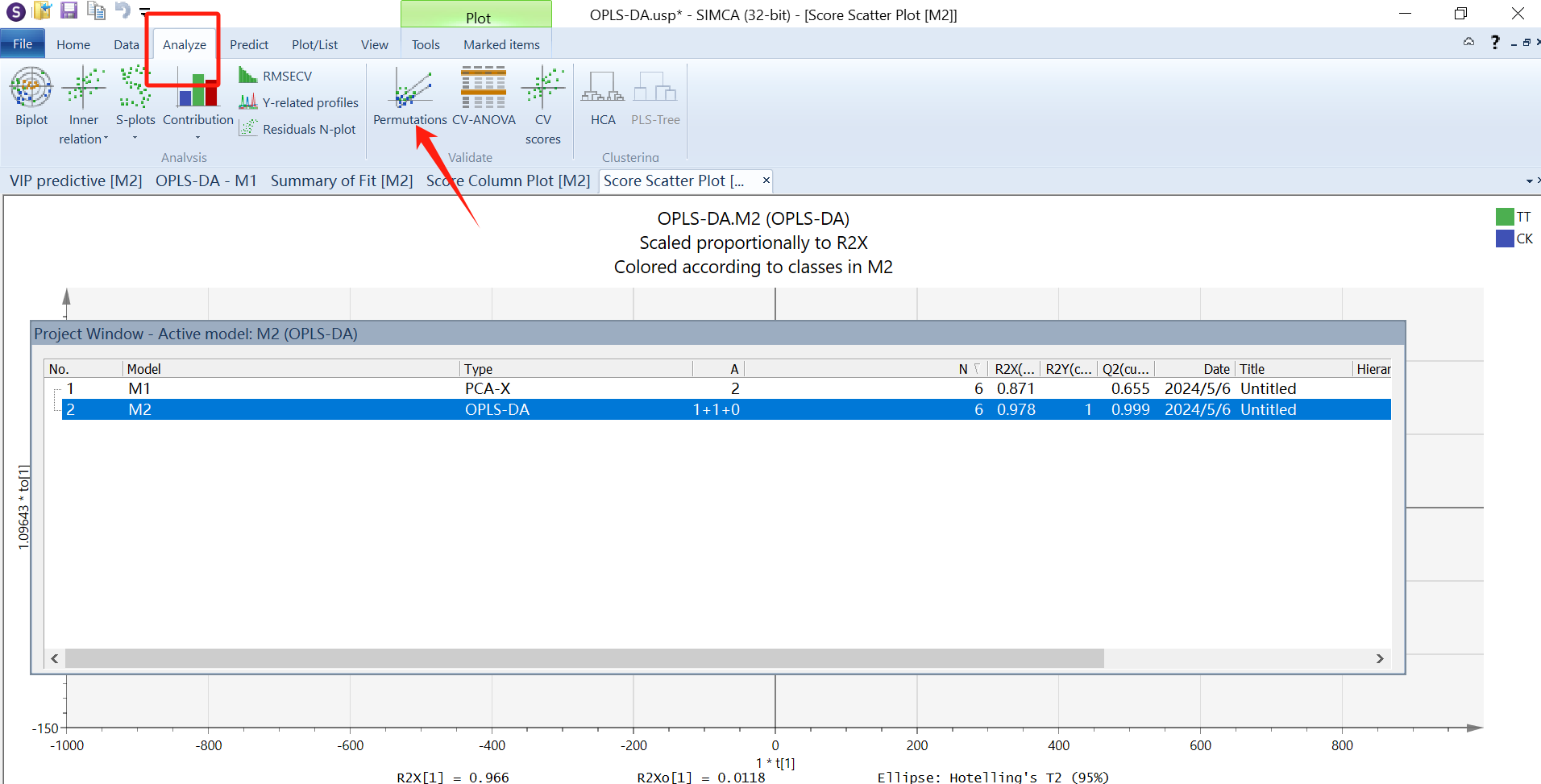 | 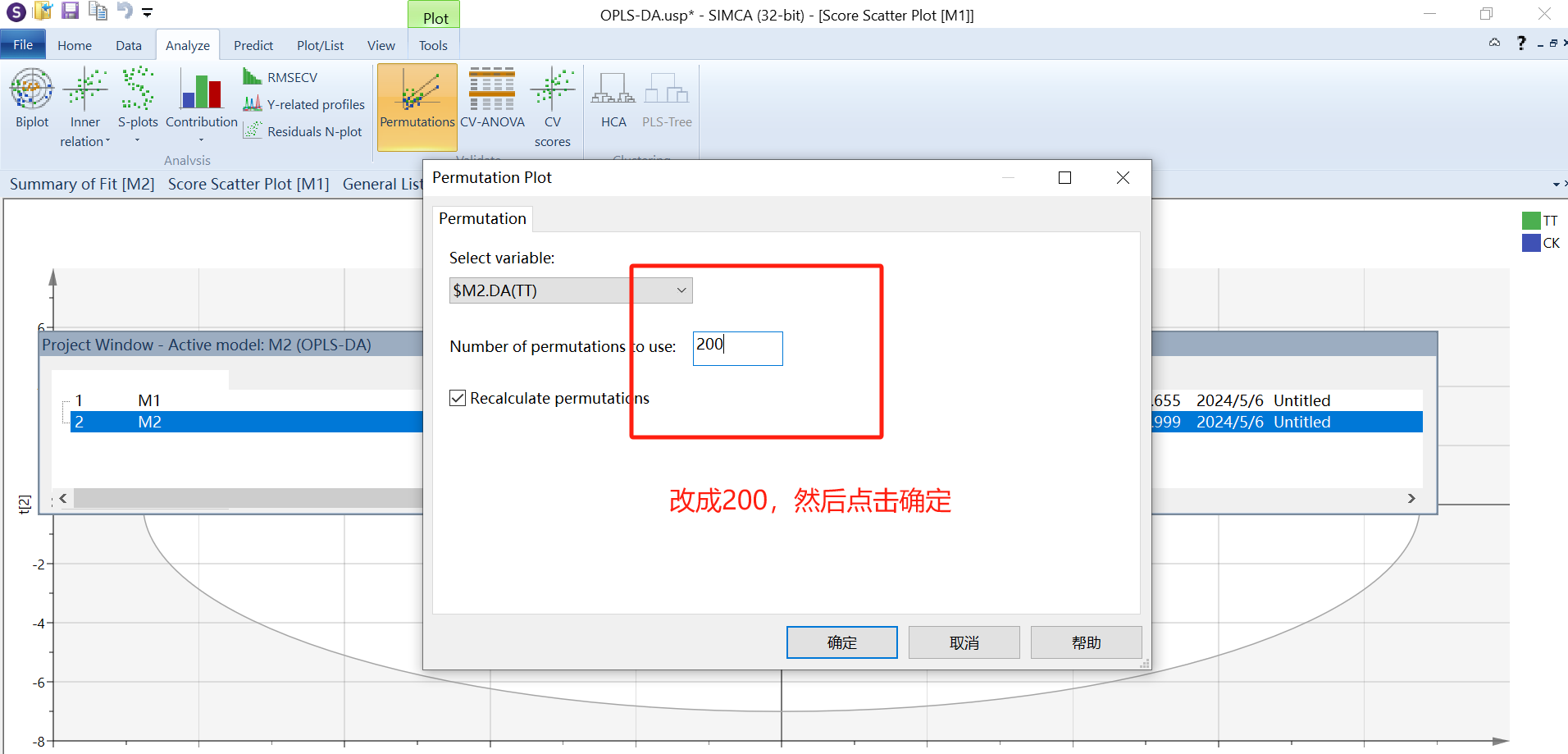 |
 | |
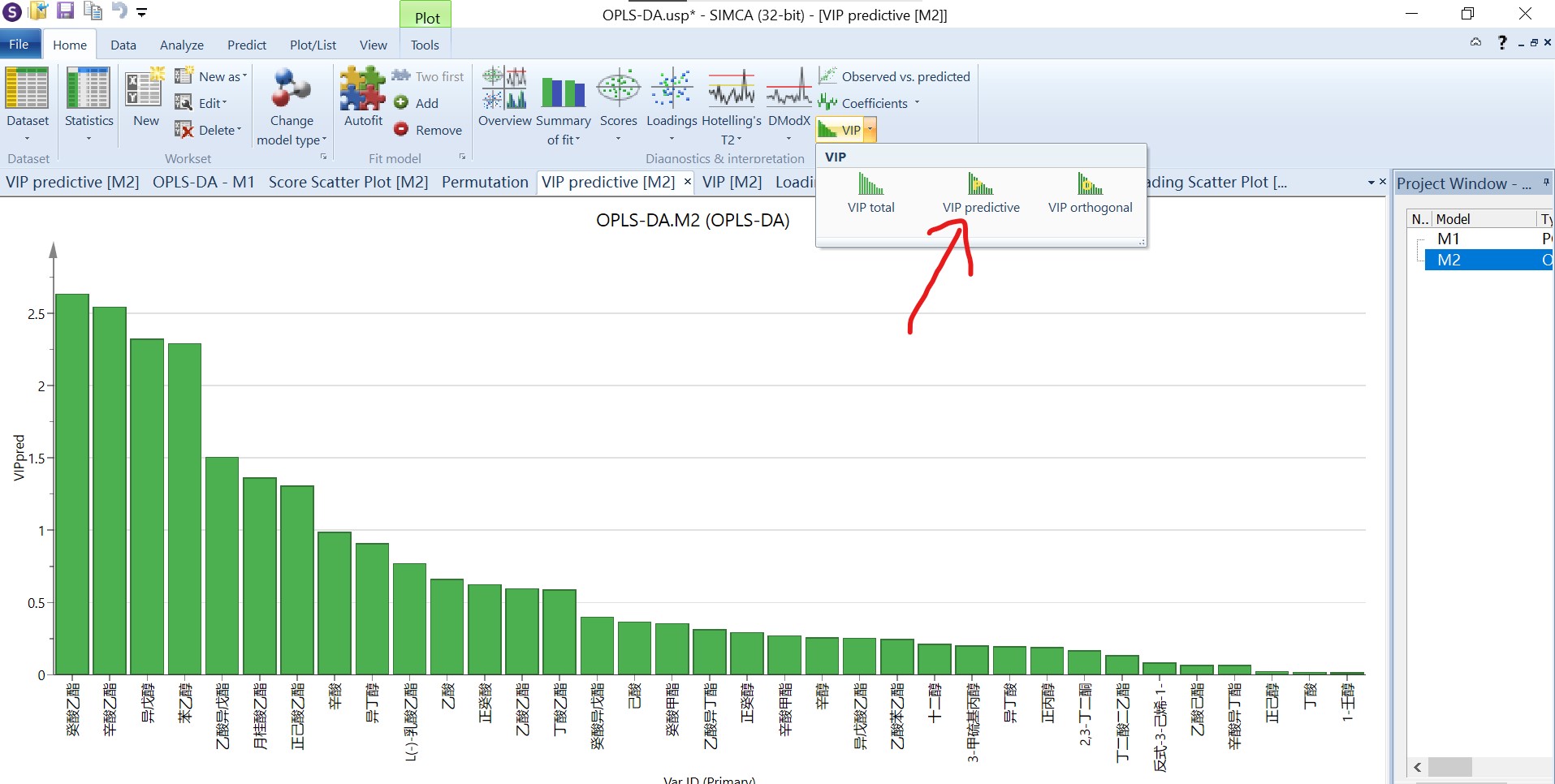
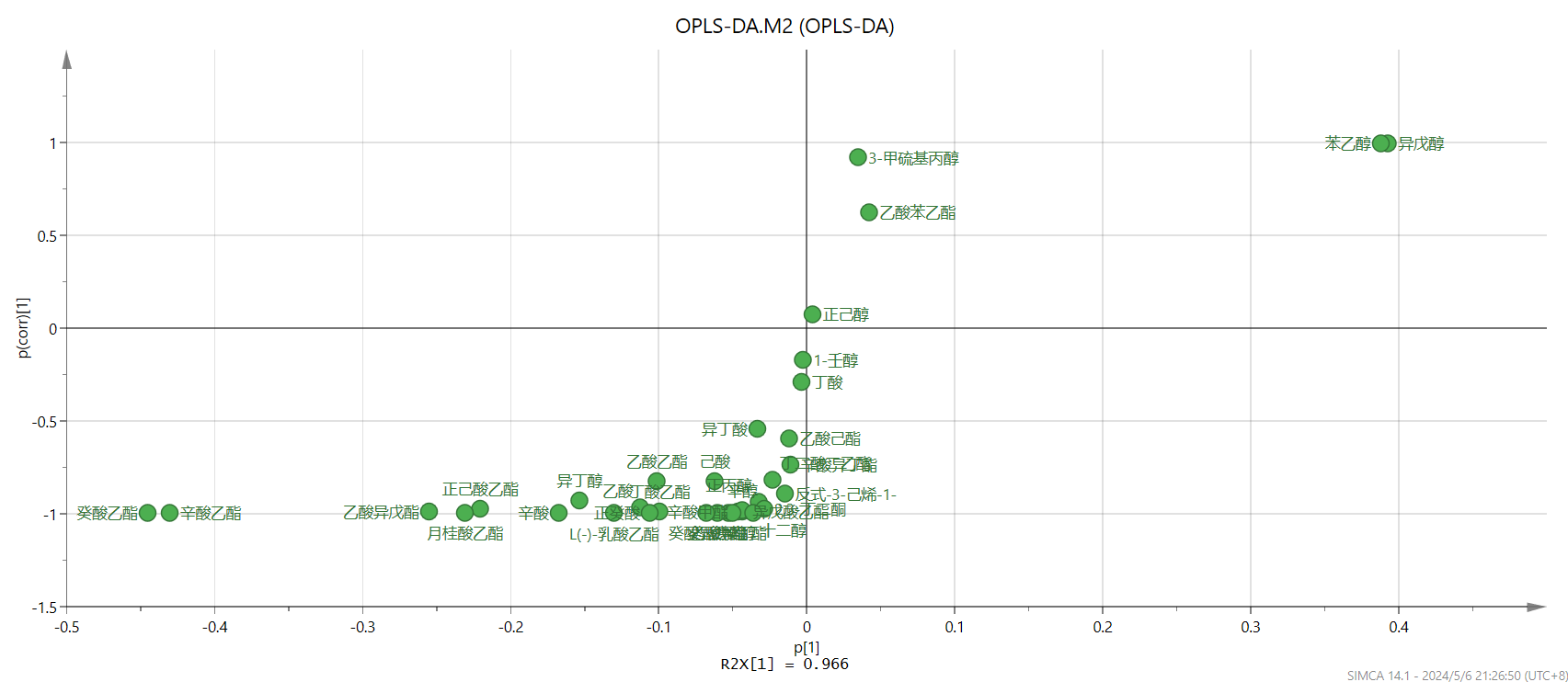
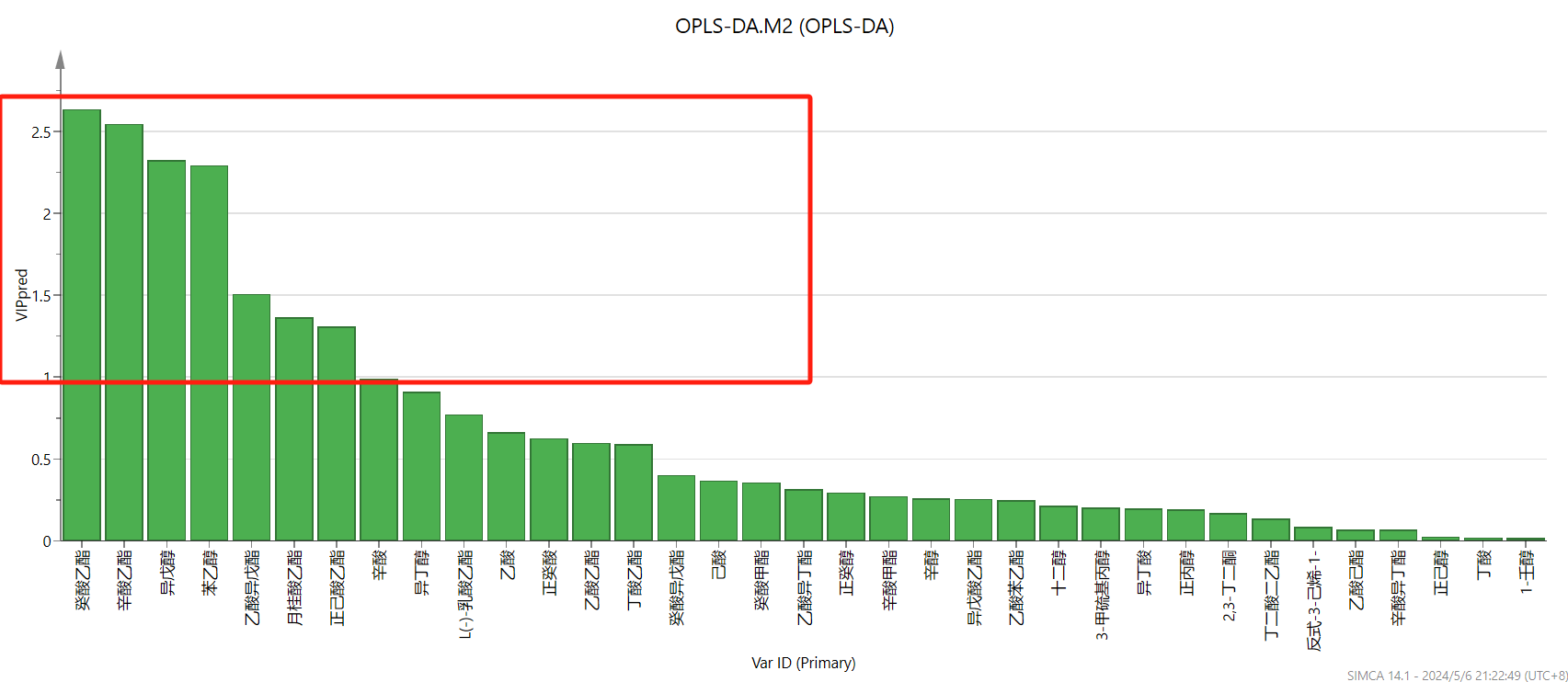
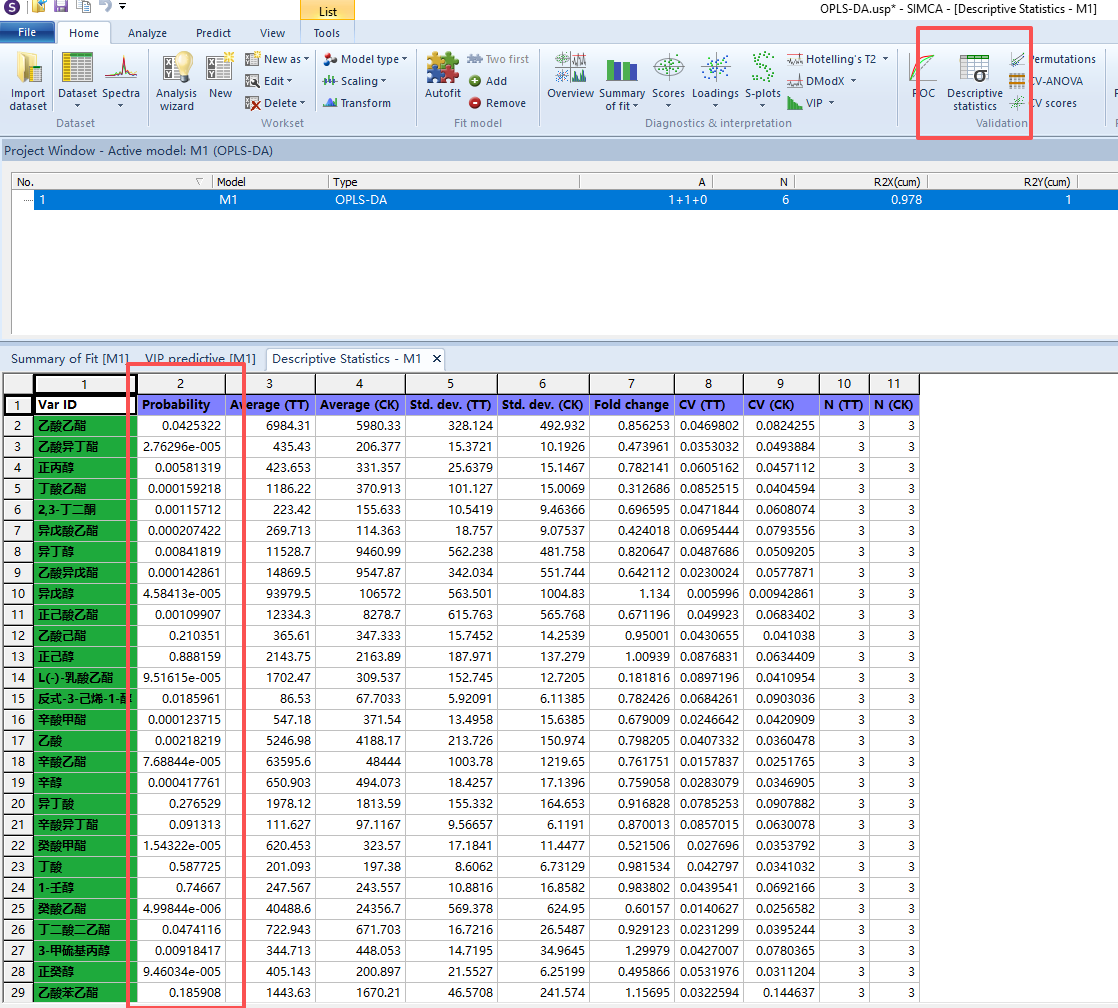
最后计算“VIP”值,“home”栏点击“VIP”旁边小三角,选择“VIP Predictive”(PLS-DA只有VIP total,OPLS-DA都有,但要用VIP Predictive),右键“create”→“list”可导出数据到origin等软件中重新作图。VIP值越大,代表该物质对于区分两组所具有的贡献越大,一般认为,VIP值大于1的代表这种物质的组间差异显著。如果想进一步提取p<0.05的差异成分,这里可以利用插件omics计算(参考SIMCA14.1的基本操作(包含p值、vip值、置换检验等,插件下载链接: https://pan.baidu.com/s/1JJouwcYVulklO0CMW0VorQ?pwd=9527 提取码: 9527
),同时获得FC值(可以用来绘制火山图),或者导出数据使用spss的独立样本t检验来逐一计算p值,需要注意这两种方式都需要进行p值校正(FDR校正)。
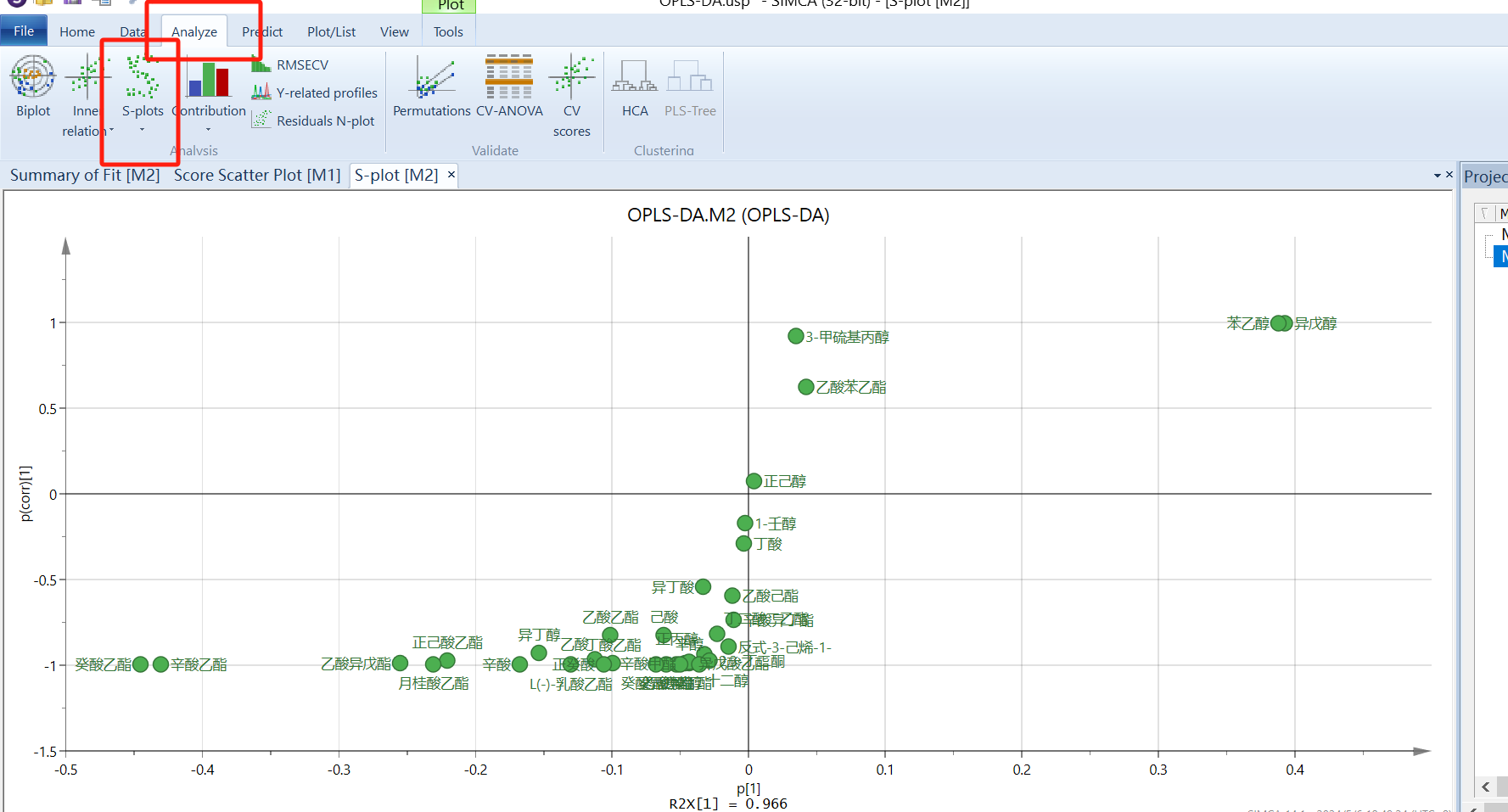
“s-plot”图的横坐标表示主成分与各类成分的协相关系数,纵坐标表示主成分与各类成分的相关系数,越靠近右上角和左下角的成分其差异越显著,其作用和VIP值图相同,二者选其一就行。
 |  |
 |  |
 |
|
结果图片展示
下图分别为s-plot图,载荷图,得分图、VIP值和置换检验图(大家可以单击右键在Format plot中对图片进行美化),一般论文只需放后三张图片即可。
 |  |
 |  |
 | |
通过OPLS-DA模型进行分类预测
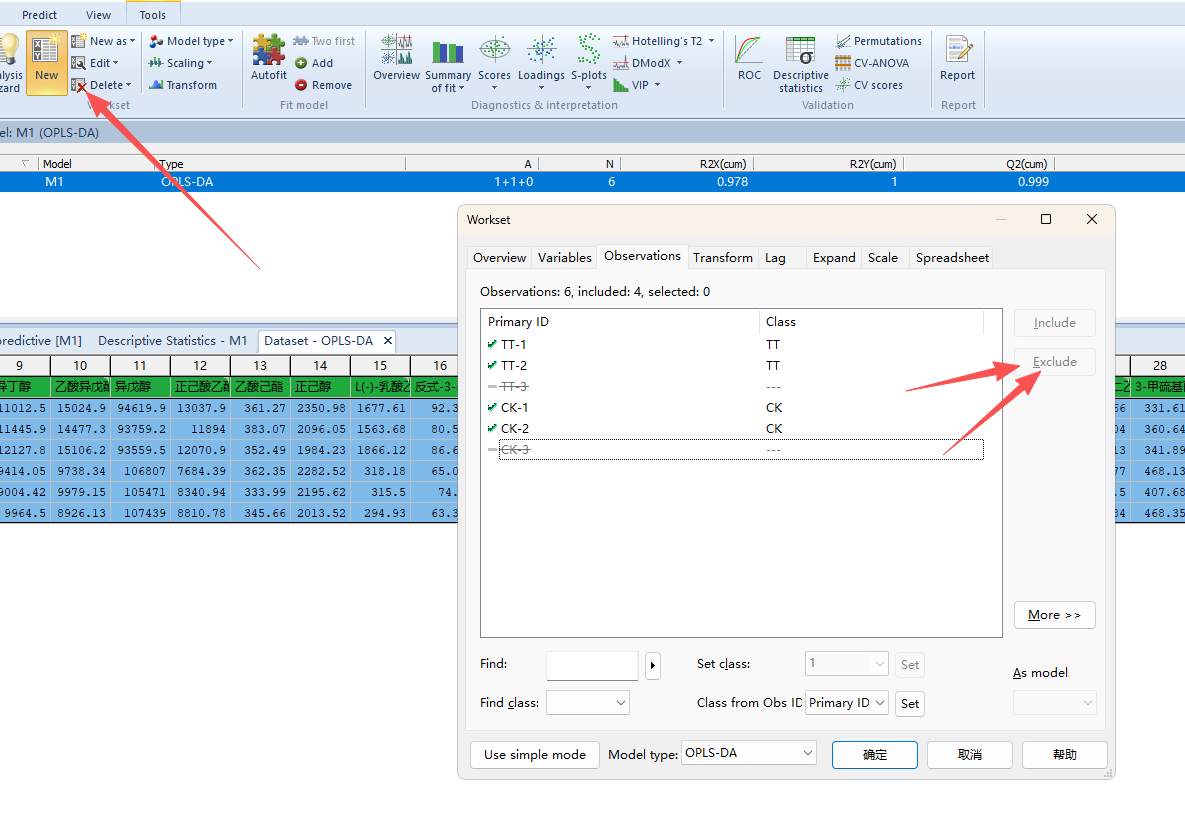
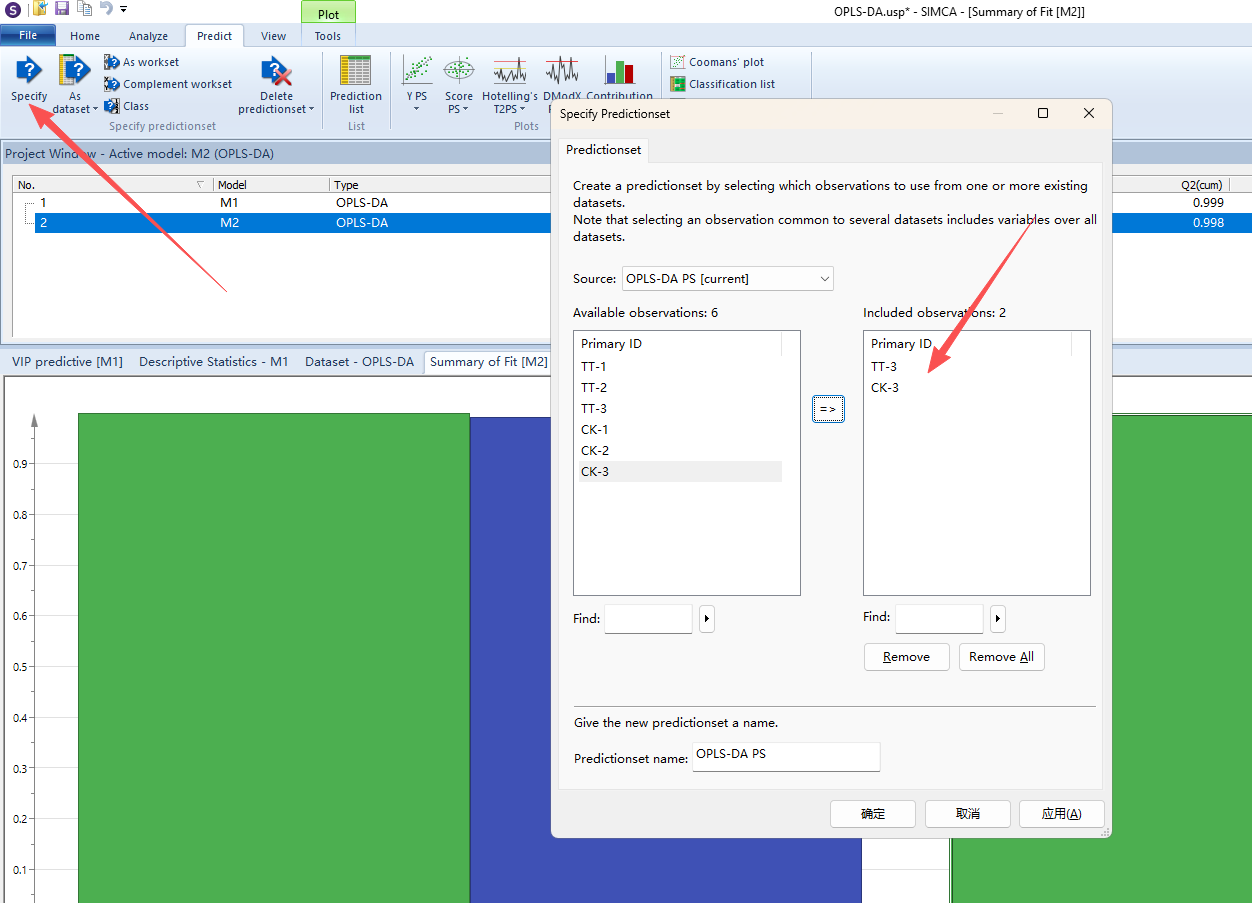
OPLS-DA除了能对现有分组数据进行差异化分析外,还能根据现有数据集进行分类预测,判断未知量是否属于现有分类组别,从而应用于产品分级、品种鉴定、产地溯源等分类判别场景。还是以上文数据为基础,先确定训练集与测试集,将TT-3与CK-3划为测试集,其余为训练集,那么我们就是以TT-1、TT-2、CK-1、CK-2为训练集进行OPLS-DA建模,然后用测试集来验证模型的预测能力。操作如下图所示,模型分类的正确率为100%,可以说明模型的预测能力较好,当然我们的数据量本身较少,存在假阴性,实际意义不大。因此OPLS-DA模型的训练集数据每组的样本量应该>30,这样的模型才比较可靠。
 |  |
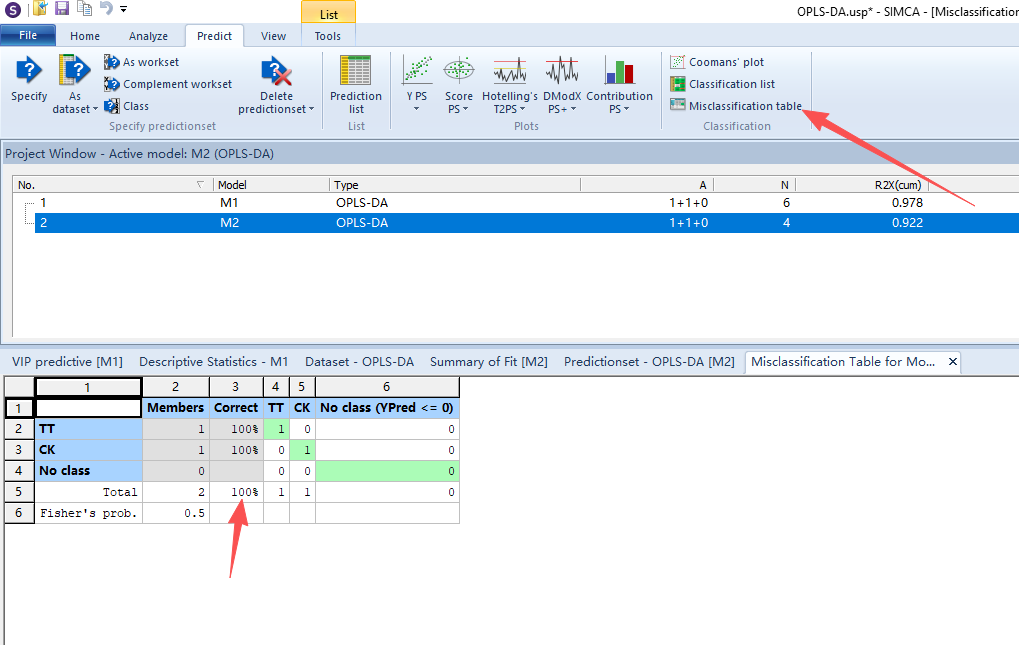 | |
以上就是全部内容,如有疑问,欢迎大家在评论区交流讨论!
教程制作不易,看完点个赞鼓励下博主吧!

参考资料:
精工致善丨多元统计分析之PCA、PLS-DA、OPLS-DA
代谢组学研究的十大误区——误区十 OPLS-DA模型能将两组分开即表示两组之间有差异?
邵淑贤,徐梦婷,林燕萍,等.基于电子鼻与HS-SPME-GC-MS技术对不同产地黄观音乌龙茶香气差异分析[J].食品科学,2023,44(04):232-239.
李少辉,赵巍,刘松雁,等.SDE-GC-MS结合OPLS-DA分析不同生态区谷子品种香气特征[J].中国农业科学,2023,56(13):2586-2596.




没有permutations那一项是什么原因
是没有permutations这个按钮选项,还是permutations不能运行?